作者:卢平
编辑:Yuki
对于 深海鱼类,很多人大概都会有 “ 随便长长就好了”“ 反正黑咕隆咚看不见”的刻板印象。 但最近发表在《科学》杂志上的一篇论文指出, 一些深海鱼类不仅能看见,而且很可能有分辨颜色的能力 ;更出人意料的是,它们这种色觉的实现机制和我们的还不太一样 [1] 。

(各种深海鱼类的世界,可能不是这样黑白打印的哦。图片来源: Wikimedia Commons.)
脊椎动物的“数码相机”
生理学知识告诉我们,脊椎动物拥有着地球上最为先进的视觉系统之一,鱼类也不例外。我们的眼睛差不多是一台高清晰度的数码相机:在眼球的前方,角膜保护下的晶状体就是一个凸透镜镜头,把进入瞳孔的光线折射到眼球后方;随后,这束描绘了我们视野中所有事物的光线,在眼球后壁的视网膜上从光信号变成了神经电信号,传入我们的大脑进行进一步的加工。
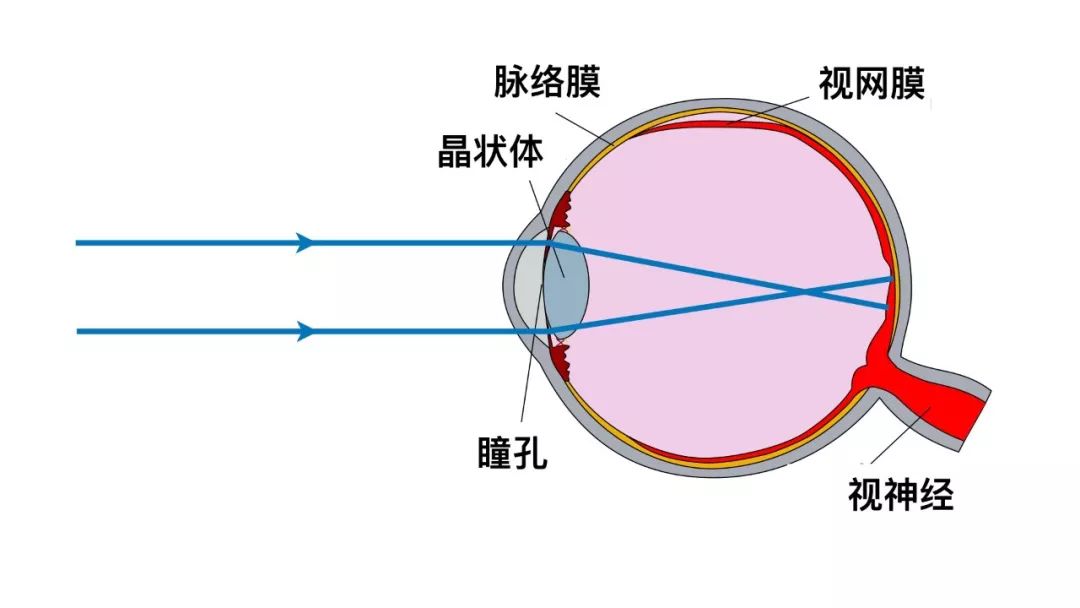
(光路通过人眼的示意图。Erin Silversmith|Wikimedia Commons. 汉化:卢平)
在数码相机中,从光信号到电信号的转换靠的是电容或者半导体感光阵列;而在脊椎动物的视网膜上, 数千万个长长的感光细胞是完成这步转换的关键 。在每个感光细胞的“头部”,有层层叠叠的膜结构,其中就嵌着视蛋白。闯进这片“丛林”的光子击打到视蛋白内部的小分子视黄醛,使其分子结构发生改变,随之引发一系列生化反应,最终关闭了细胞膜上的离子通道。被“堵”在细胞外、带正电荷的钠离子不断积累,让感光细胞内外的电压差越来越大,激发了像多米诺骨牌一样沿着细胞膜向前推进的“离子人潮”,也就是神经电信号,由感光细胞向后续的神经元依次传递,奔向视神经的深处。

(视网膜的组织结构分层。OpenStax College|Wikimedia Commons. 汉化:卢平)
这套“光电元件”的核心就是视蛋白。不同的视蛋白在基因组中由不同的基因序列来编码,对不同波长(也就是“颜色”)光线的敏感度也不一样。 在脊椎动物的视网膜上,有两类主要的感光细胞 :视杆细胞的头部是个长长的圆柱体,所含的视蛋白是视紫红质(rhodopsin),对蓝绿光最为敏感。视锥细胞的头部则是锥形,每个视锥细胞含有一种对蓝光、绿光或者红光敏感的视蛋白。
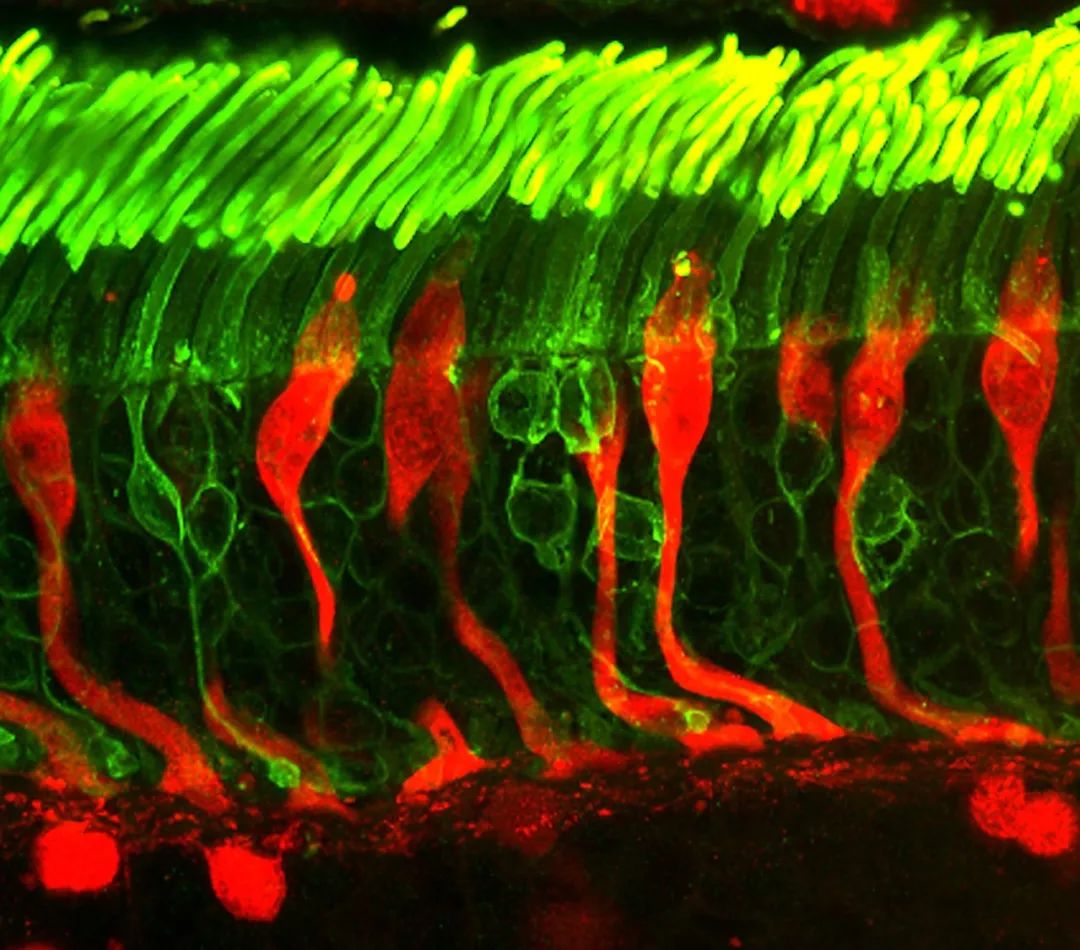
(荧光显微镜下的视杆细胞(绿色)和视锥细胞(红色)。图片来源:National Eye Institute / Flickr.)
显而易见,两类感光细胞在视觉形成上有着不同的分工: 对于有色觉的脊椎动物来说,视锥细胞就是多彩世界的来源 ——不同的视锥细胞分别采集红、绿、蓝等光线,合成出各种我们感知到的色彩。但是,视锥细胞对光线的敏感性比较差;而我们眼睛里大部分的感光细胞其实是视杆细胞——它们对光子的探测十分灵敏。 在夜间或者昏暗环境下,我们看到的没有色彩的图像基本都是视杆细胞的贡献 。
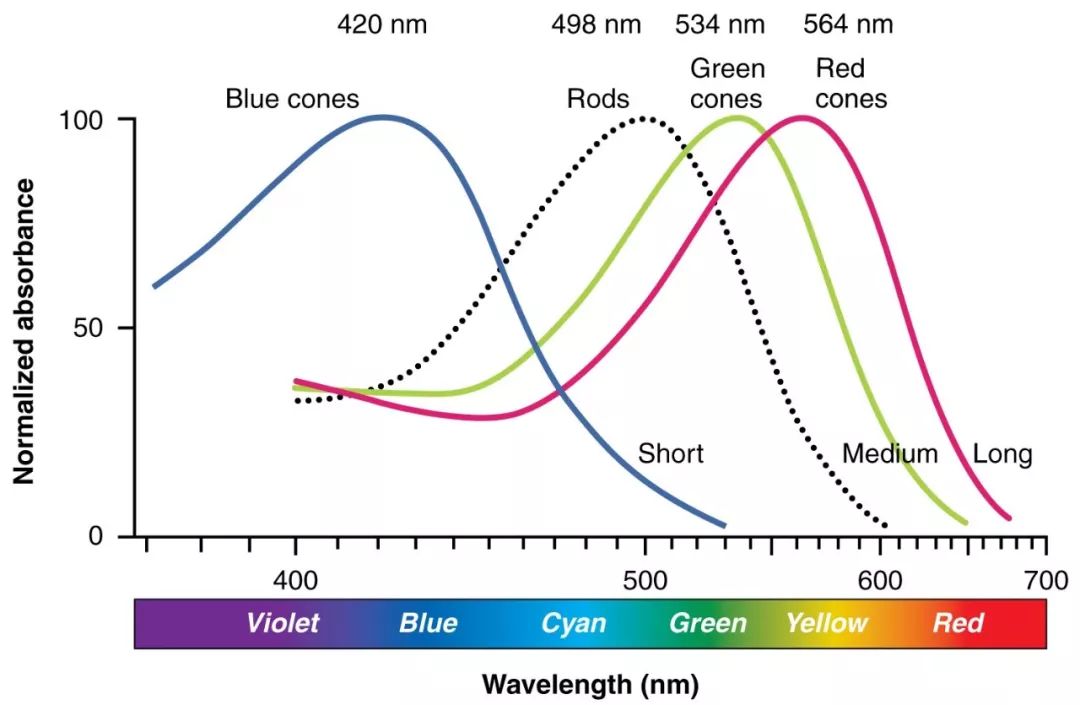
(从左到右分别是三种视锥细胞(彩色)和视杆细胞(虚线)的光敏感度曲线,X轴是波长也就是颜色,Y轴是吸收强度。图片来源: Wikimedia Commons.)
所以,视杆细胞只有一类,主要负责感光,跟分辨颜色没关系;而视锥细胞提供彩色视觉。视锥细胞种类少的物种,能辨别的颜色就少,比如鸟类有四种视锥细胞,而大部分哺乳类只有两种。
至少,教科书上是这么写的。
是升级改造,还是放弃治疗?
这么精巧的视觉系统,对于很多动物来说是捕食逃命、趋利避害的必需品,在适应性演化的过程中也无疑受到了“特别关照” 。比方说,生活在东南亚的眼镜猴,就有着和自己大脑重量相当的大眼睛,视网膜的面积和视杆细胞的密度都十分惊人,以便在夜间的雨林里捕捉昆虫。相反,恰恰也是因为精巧而耗费发育能量,视觉系统一旦弃之不用又很容易退化消失,例如生活在地下的裸鼹鼠、钩盲蛇和生活在洞穴里的鱼类。

(眼睛退化的墨西哥丽脂鲤。H. Zell|Wikimedia Commons.)
深海同样是个“暗无天日”的世界。在海面二百米以下的微光层(twilight zone),从水面照射下来的光线已经所剩无几,更不可能支撑植物的光合作用。然而,这里仍是生命的乐园——从上层海水中随机掉落的生物残骸,能在微光层得到有效的“回收利用”。
很多浮游生物还有着奇特的垂直迁徙行为,白天下潜到微光层躲避捕食者,晚上则到上层海水中“上夜班”填饱肚子。 为了适应微光层的黑暗世界,很多浮游生物演化出了生物荧光,用于吸引猎物、识别同类和谈恋爱。

(很多磷虾物种就是微光层的常客。Uwe Kils, CC BY-SA 3.0, Wikimedia Commons.)
那么, 生活在黑暗大洋中的鱼类,它们的视觉系统又有着怎样的适应呢?是经过了“魔改”还是干脆放弃呢? 为了弄明白这个问题,来自欧洲和澳大利亚的研究者收集了一百多种鱼的基因组序列数据,其中包括几个生活在深海的物种,想看看这些深海居民的视蛋白基因有没有什么特殊之处。
(外形奇特的鞭尾鱼,双眼突出向上,英文叫“管子眼”(tube-eye)。J. F. Hennig, Public Domain|Wikimedia Commons.)
“对不起,基因多就是可以为所欲为”
人比人气死人,鱼比鱼发论文。大部分鱼类和其它脊椎动物一样,只有一种视杆细胞,用一个RH1基因来编码唯一一种视紫红质蛋白。但是,有四种深海鱼类让科研人员吃了一惊:冰底灯鱼( Benthosema glaciale )有五个不同的RH1基因,鞭尾鱼( Stylephorus chordatus )有六个,而两个银眼鲷科的物种——短鳍拟银眼鲷( Diretmoides pauciradiatus )和银眼鲷( Diretmus argenteus ),则分别有18个和38个RH1基因的拷贝!
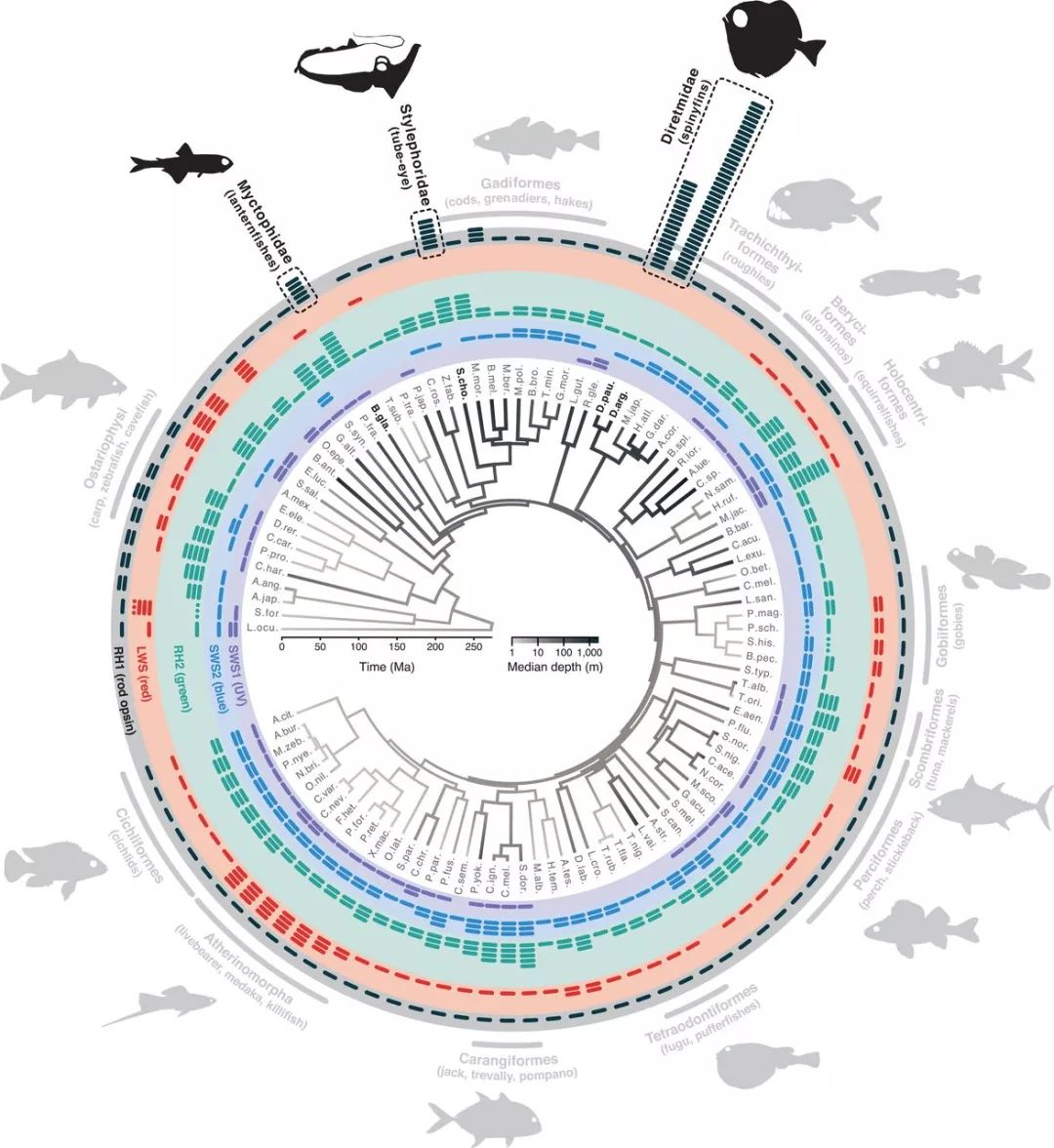
(一百多种鱼类的演化关系树,黑色标出的就是四种有5个以上RH1基因的物种。图片来源:参考文献[1])
这种多拷贝现象要归结于一种名为 基因复制 (gene duplication)的一种变异事件。此类事件在基因组中倒是并不罕见,但是在演化历史中,大部分复制产生的基因序列都会逐渐“退化”,丧失合成蛋白质的功能。所以,这么多RH1基因拷贝,也不见得都能正常合成出堪用的视紫红质蛋白。于是,研究者们又解剖得到了这些鱼类的视网膜,从中提取了基因序列合成蛋白质的中间环节——RNA分子进行测序分析。结果表明,对冰底灯鱼和近亲瓦式角灯鱼来说,有三种不同的视紫红质在视网膜中得到了合成,鞭尾鱼有五种,而对于成年的银眼鲷个体来说,这个数字则有14种之多。

(银眼鲷真容。图片来源:Emma Kissling, Public Domain|Wikimedia Commons.)
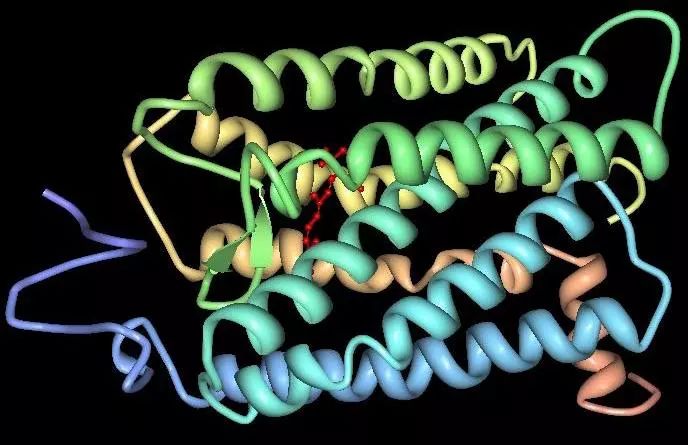
(牛的视紫红质蛋白三维结构,注意中间那个红色的小分子就是视黄醛。图片来源:Palczewski et al, Science, 2000.)
所谓“不同的视紫红质”,说的是这些蛋白“变种”的氨基酸序列发生了变化,而作为一个视蛋白,序列变化很可能会导致“敏感区”不同。果不其然,通过在体外合成银眼鲷的视紫红质,研究者发现这些不同的变种分别对不同波长的光线敏感,范围一直从447纳米的蓝光到513纳米的黄绿光——这意味着, 银眼鲷视杆细胞里的这些视紫红质变种,可能像其它脊椎动物视锥细胞中的各种视蛋白一样,能够区分颜色 。
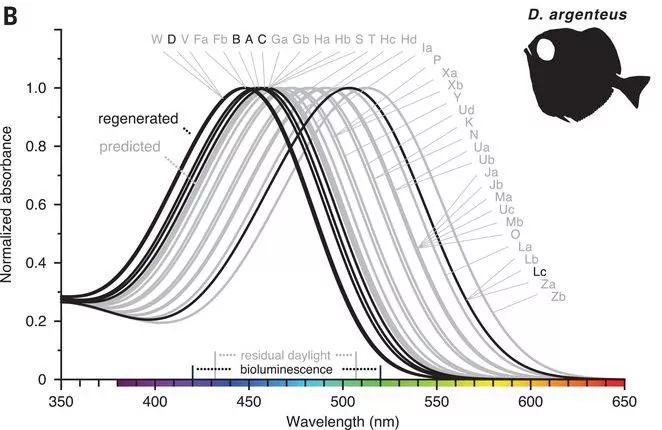
(银眼鲷不同视紫红质的光敏感度曲线,X轴是波长也就是颜色,Y轴是吸收强度。图片来源:参考文献[1])
当然,研究做到这里,我们仍不能确定地说“银眼鲷的视杆细胞能形成彩色视觉”,这需要行为学的实验来进一步验证。不过,银眼鲷的视杆细胞所跨越的447-513纳米这个色彩区间,恰好是在微光区最常见的、偶尔穿越上层海水的光线波长,因此拥有对蓝绿光敏感的视紫红质对于这些深海鱼类来说很可能是有利的;而这个区间也是各种浮游生物的荧光波长所在,这么看来能够区分不同的蓝和绿就更有意义了。
另外,分子序列演化模型也表明, 银眼鲷的这三十多个RH1基因,经历了百万年之久的“正向选择” ——也就是说,这些视紫红质变种的出现,很可能帮助银眼鲷完成了对微光层环境的适应,在黑暗中看到了五彩斑斓。

(一些两栖类也是有两种视杆细胞的。图为中华蟾蜍。图片来源:DrewHeath|Wikimedia Commons.)
2004年,有研究指出一种沙漠守宫没有视杆细胞,但却能在月光下用视锥细胞分辨颜色 [2] ;2017年,研究者发现蛙类的两种视杆细胞能在极微弱的光线下分辨蓝色和绿色 [3] 。 充满了随机性的演化永远是“将计就计”“见招拆招”,造就了适应环境而又千奇百怪的亿万物种 。
2019年的“世界生物多样性日”刚刚过去。正如大学老师曾告诉我们的那样——生物学科的最重要规律,恐怕就是“所有生物学规律都有例外”。这些例外不仅丰富着我们的生物课本,也可能意味着新的生物医学突破,新的生态治理办法等等。这是生物多样性的体现,是生命在三十八亿年中从不停歇的脚步回响。
作者名片

参考文献
[1] Musilova, Zuzana, et al. Vision using multiple distinct rod opsins in deep-sea fishes. Science 364.6440 (2019): 588-592.
[2] Roth, Lina SV, and Almut Kelber. Nocturnal colour vision in geckos. Proceedings of the Royal Society of London. Series B: Biological Sciences 271.suppl_6 (2004): S485-S487.
[3] Kojima, Keiichi, et al. Adaptation of cone pigments found in green rods for scotopic vision through a single amino acid mutation. Proceedings of the National Academy of Sciences. 114.21 (2017): 5437-5442.



 科普中国公众号
科普中国公众号
 科普中国微博
科普中国微博

 帮助
帮助