在现代生物技术和医药研究中,蛋白质工程扮演着至关重要的角色。通过修改蛋白质的氨基酸序列,蛋白质工程可以改善或赋予蛋白质新的生物化学性质,如增强酶的催化效率、提高药物的亲和力或改善其热稳定性。这些改进对于开发新药、治疗疾病以及提高生物制造的效率等方面都是非常关键的。
蛋白质工程需要从数以万计的候选突变体中筛选出最优突变体,其中的有利突变是指那些能够改善蛋白质某一或多个生物化学属性的遗传变异,增强蛋白质的稳定性、亲和力、选择性或催化效率,使其更适合特定的应用。然而,通过实验验证高适应性突变体的成本大、时间长,此外,多个有益突变的组合往往会受到负表观遗传效应的影响,使得蛋白质的功能因突变而降低,这些因素都在不同程度上增加了高效蛋白质设计的复杂性。
近几年,基于深度学习的预测与筛选方法在实际应用中得到了验证与应用:通过分析大量数据,学习蛋白质序列、结构与功能的关系,能够提高蛋白质设计的准确性和效率。但多数方法是基于多序列比对 (MSA) 或蛋白质语言模型 (PLM) 对蛋白质序列进行特征的提取,存在诸多局限,例如依赖多序列比对的质量,受限于同源信息;或需大量数据和复杂模型,训练成本高。此外,直接应用预训练模型到新任务,对模型的泛化能力和表达能力更是一大挑战。
为此,上海交通大学洪亮课题组研发了一种名为 PROTLGN 的微环境感知图神经网络,能够从蛋白质三维结构中学习并预测有益的氨基酸突变位点,指导具有不同功能白质单位点突变和多位点突变设计,超过 40% 的 PROTLGN 设计单点突变体蛋白质优于其野生型对应物。成果已发表在 JCM。

PROTLGN :轻量级图神经去噪网络的搭建
PROTLGN 框架:基于图神经网络的蛋白质学习网络
PROTLGN 是一种基于图神经网络的蛋白质表征学习模型,其核心架构如下:
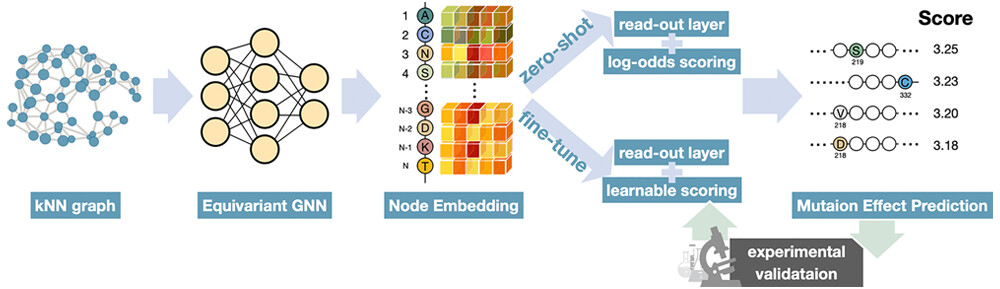
PROTLGN 架构
kNN 图 (k-Nearest Neighbors Graph):
输入蛋白质的氨基酸残基作为图中的节点,通过 k 临近算法确定边基与氨基酸残基之间的空间距离,进而构建蛋白质的拓扑结构,为后续的图神经网络处理提供基础。
等变 GNN (Equivariant Graph Neural Network):
在三维空间中,蛋白质的结构可能会发生旋转或反射。等变 GNN 作为核心网络层,设计成能够识别并保持这种旋转不变性的结构,即无论蛋白质图形如何旋转,网络的输出对于相同的蛋白质结构都应该是一致的。
节点嵌入 (Node Embedding):
在图表示的蛋白质中,每个氨基酸残基被表示为图中的一个节点,以便于机器学习模型能够捕捉和理解节点之间的复杂关系。
输出层与得分 (read-out layer & score):
利用等变 GNN 学习到的节点表示识别有益的突变位点,预测突变对蛋白质功能或结构的潜在影响。同时作为模型的最后一层,将预测结果转为量化评分。
验证 (Validation):
使用实验生物学方法,如酶联免疫吸附试验 (ELISA)、差示扫描荧光热稳定性分析 (DSF) 等对模型预测的突变体进行实验验证,测试其生物学功能。
PROTLGN 的训练过程:训练-预测-微调
PROTLGN 的训练过程如下图所示,其中包含训练、预测和模型微调:
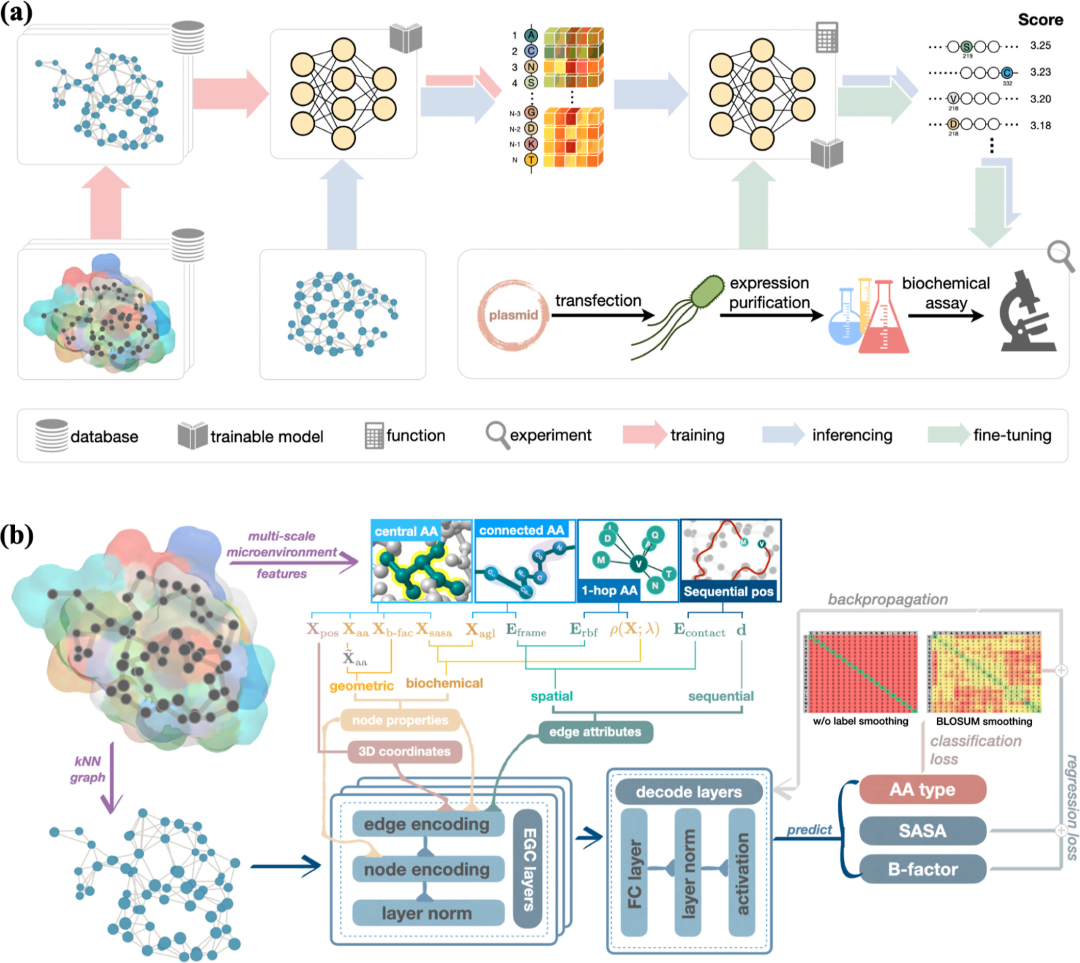
PROTLGN 预训练和预测流程
自监督预训练 (Self-supervised Pretraining):
PROTLGN 首先在野生型蛋白质上进行自监督预训练,任务是氨基酸类型的去噪 (AA-type-denoising)。
输入图中包含的三维坐标信息是节点属性的一部分,用于更准确地表示氨基酸残基在蛋白质三维空间中的位置。
三维坐标信息与氨基酸的物理和生化属性(如氨基酸类型、SASA、B-factor 等),共同构成了输入图的节点和边的属性。这些属性被用来构建 KNN 图,其中每个节点(氨基酸残基)根据其与其它节点的空间距离相互连接。
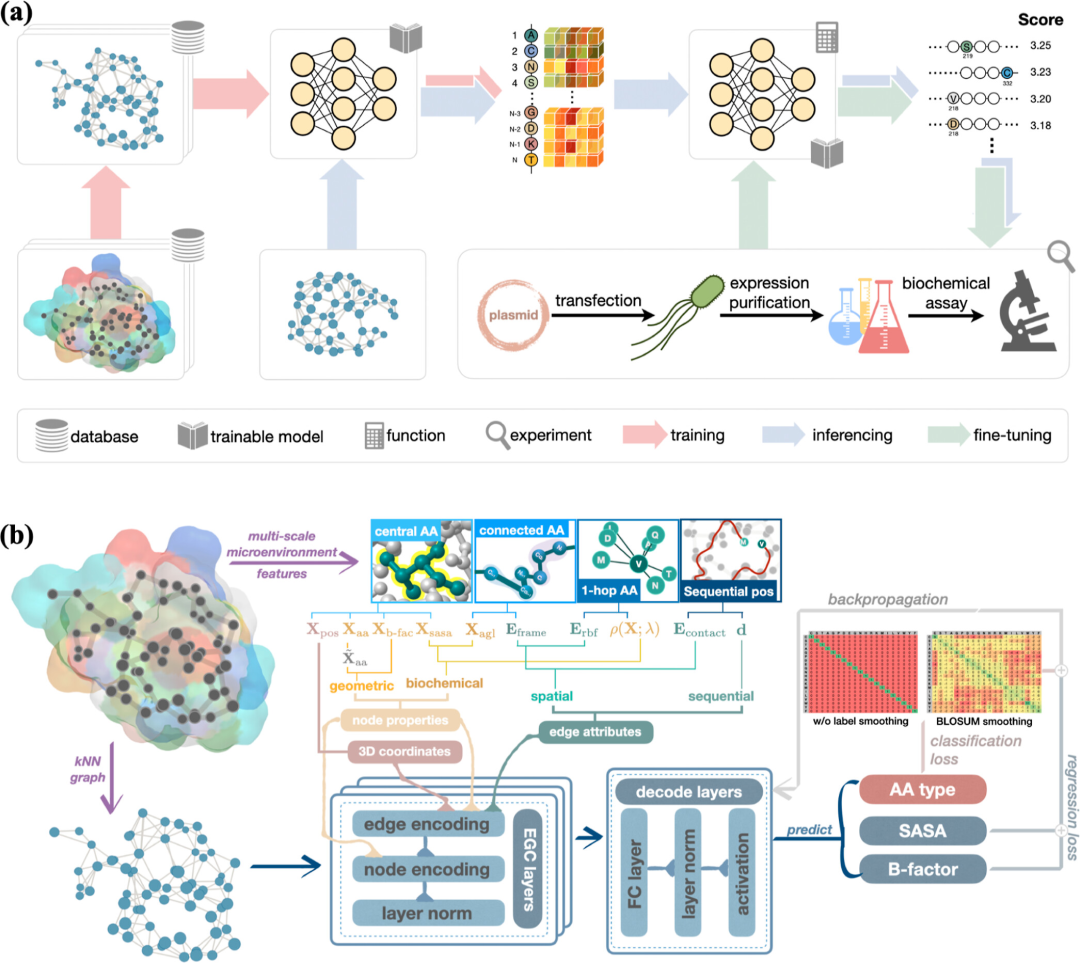
PROTLGN 的自监督学习过程
等变图卷积层 (EGC):
预训练中使用等变图神经网络 (equivariant graph neural networks, EGC layers),负责处理输入的蛋白质图,通过本层,模型能够学习到在旋转和平移变换下保持不变的节点嵌入,帮助处理不同蛋白质的结构。
EGC 层是图神经网络的核心,能够处理图结构数据,并且保持对蛋白质空间结构变化的敏感性,这对于理解蛋白质的三维结构至关重要。
在自监督学习过程中,EGC 层接收具有噪声的野生型蛋白质图作为输入,并输出节点的嵌入表示,这些嵌入表示考虑了氨基酸残基之间的空间关系。
噪声注入 (Noisy Input Attributes):
在训练过程中,对野生型蛋白质的输入属性注入噪声,模拟自然界中的随机突变。
零样本预测 (Zero-shot Prediction):
蓝色箭头表示当考虑蛋白质突变时,模型使用预训练阶段学到的知识来预测突变对蛋白质功能可能产生的影响。
湿实验评估 (Wet Biochemical Assessments):
将突变体的预测与湿实验评估相结合,可以更新预训练模型,以更好地适应特定蛋白质和功能。
微调 (Fine-tuning):
图示绿色箭头部分,结合湿实验的评估,预训练模型可以根据特定的蛋白质和功能进行更新和优化,提高预测的准确性和适应性。
为了进一步利用生物学的先验信息来提高模型的泛化性和表达能力,研究人员还采取了 3 个额外的措施:
对输入的氨基酸类型进行加噪,模仿自然界中的随机突变;
在氨基酸节点预测的损失函数打分机制中,引入标签平滑来鼓励同类氨基酸之间的置换;
利用多任务学习策略,让预训练模型学习多种预测目标,从而训练一个「一词多用」的图表示学习模型。
挖掘蛋白质定向进化潜能:PROTLGN 提供有效策略
为了验证 PROTLGN 对蛋白质突变体活性预测的准确性,本研究在多种蛋白质的不同生物学功能上进行了广泛的验证工作,以确保 PROTLGN 的普适性,其中包括 VHH 抗体、多种荧光蛋白(如绿色、蓝色和橙色荧光蛋白)、以及核酸内切酶 (KmAgo) 等,涵盖了热稳定性、结合亲和力、荧光亮度和单链 DNA 切割活性等蛋白质工程中常见的功能改造目标。
实验数据显示,即使在缺乏实验数据或仅有少量类似蛋白质实验数据的情况下,PROTLGN 仍能达到 40% 的单点突变成功预测率,并且在某些情况下能够同时提升多种生物学功能。
PROTLGN 与荧光蛋白:预测模型的迁移能力
研究人员采用 PROTLGN 模型,对绿色荧光蛋白 (GFP) 进行了精细调整,以开发出专门针对荧光强度优化的评分函数。从深度突变扫描 (DMS) 数据库中随机选取 1,000 个已标记的 GFP 突变体进行微调训练,从而提高了模型预测荧光强度变异的准确性。
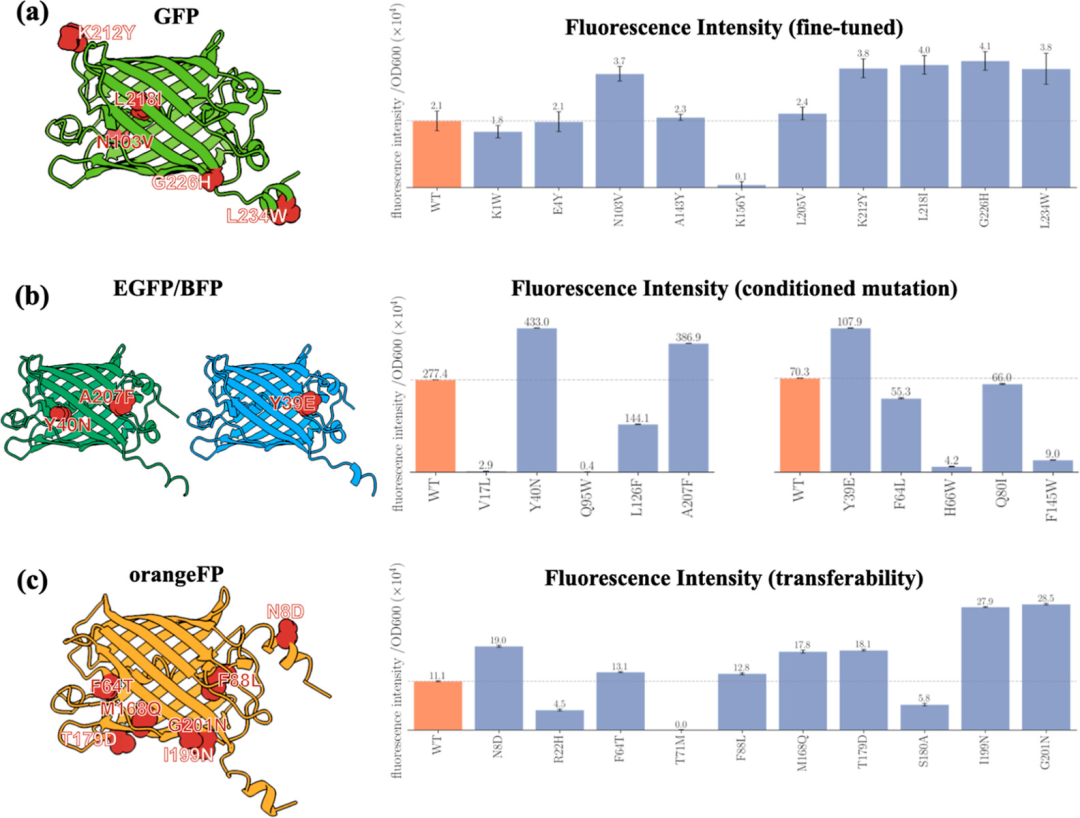
荧光蛋白实验结果
左侧展示蛋白质结构,红色球体突出显示了发生突变的氨基酸残基
右侧展示荧光强度数据,不同突变体与 WT 进行对比
图 a 评估了从少量标记的绿色荧光蛋白 (GFP) 变体中学习到的特定功能适应度评分函数的实用性。在 10 个突变体中,有 5 个展现出比野生型 (WT) 更高的荧光强度,其中表现最佳的突变体荧光强度达到了 WT 的 2 倍。
此外,该实验检验了同一评分函数在与 GFP 来自不同蛋白家族、活性区域不同、序列同源性约 21% 的橙色荧光蛋白 (orangeFP) 上的表现。研究人员利用微调后的 PROTLGN 对 orangeFP 的单点突变体进行排名,并挑选前 10 个变体进行湿实验表达和检验。
在这些突变体中,有 7 个表现出比 WT 更高的荧光强度,这一结果彰显了模型的强大迁移能力。
PROTLGN 与 VHH 抗体:零样本 PROTLGN 的性能
实验人员使用 PROTLGN 模型,在没有实验数据的情况下,通过约 30,000 个未标记的蛋白质结构进行预训练,选择具有最高适应度预测的 VHH 抗体变体中的前 10 个突变体进行湿实验评估。
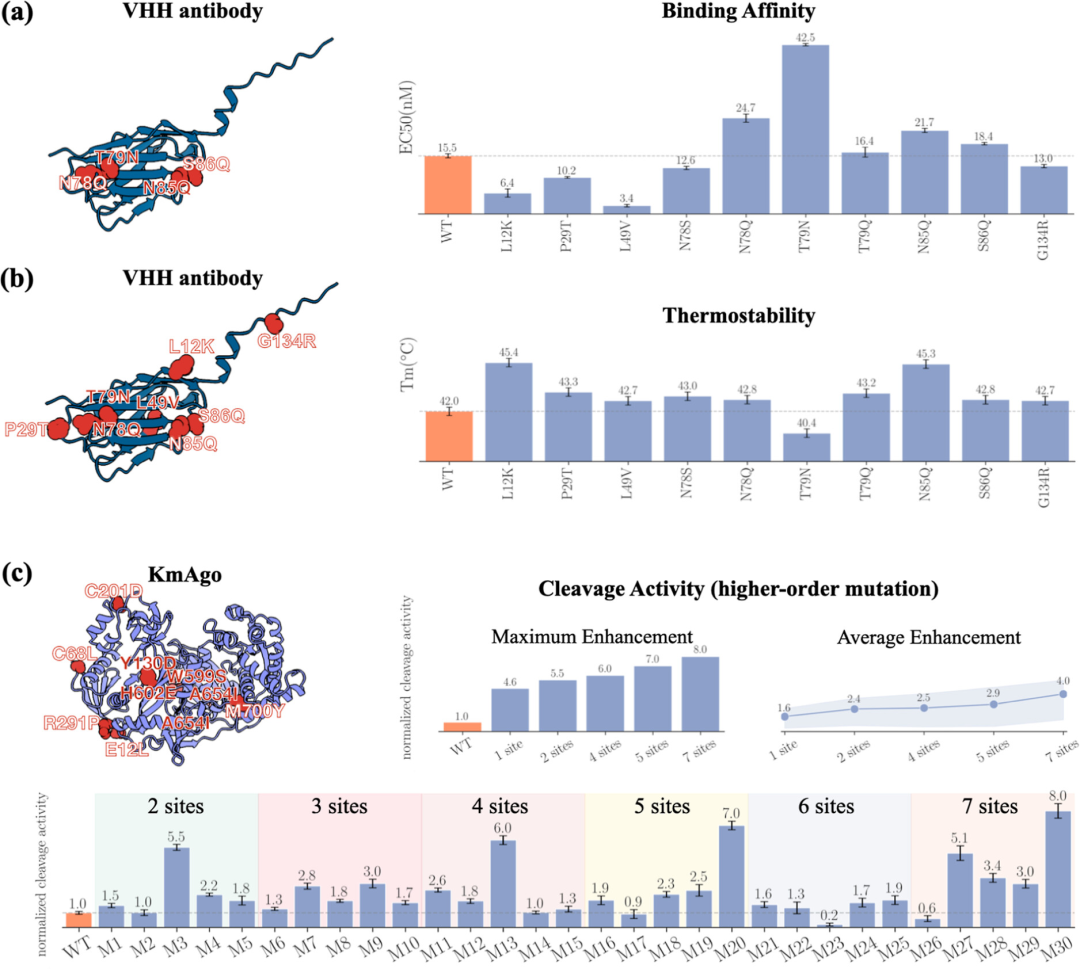
PROTLGN 设计的 VHH 抗体结果
(a):左侧展示 VHH 抗体的结构,右侧展示 VHH 抗体及其单点突变体的结合亲和力
(b):左侧展示 VHH 抗体的结构,此处不同位点产生突变,右侧展示 VHH 抗体及其单点突变体的熔点温度
有 3 个突变体在结合亲和力和热稳定性两方面都表现优异,这证实了 PROTLGN 在指导 VHH 抗体突变设计中的有效性,尤其是在提高抗体的性能方面。
PROTLGN 的自监督学习策略为蛋白质工程提供了一种强大的工具,能够在没有实验数据的情况下进行准确的突变预测。
PROTLGN 与 Ago蛋白:寻找最优单点突变组合
研究人员利用 PROTLGN 对 12 个已知的单点突变进行组合评分,筛选出 2—7 个位点的前 5 个高阶突变候选体,共 30 个突变体,以期通过湿实验评估找到性能更优的 Ago 蛋白变体。
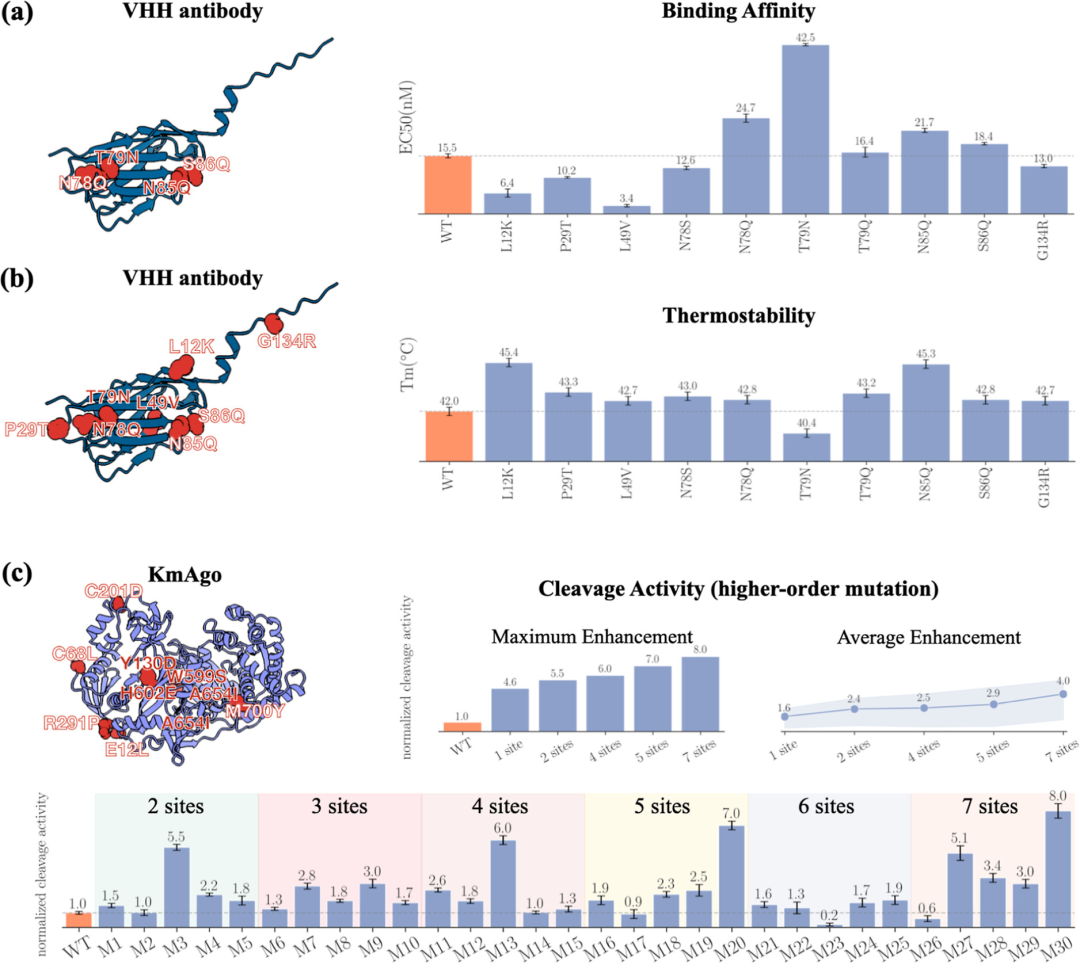
PROTLGN 设计的 KmAgo 突变体及实验结果
左上:KmAgo 蛋白的结构
右上:不同数量突变位点的 KmAgo 突变体的最佳活性。这可能表明随着突变位点的增加,活性如何变化
中下:KmAgo 及其多突变位点突变体的切割活性
实验结果显示:
活性提升:与野生型 (WT) 相比,90% 的突变体显示出增强的 DNA 切割活性。
最佳突变体:最佳的突变体是一个 7 位点突变体,其活性比 WT 高出 8 倍。
高阶突变体的优势:无论是在最大活性提升还是平均提升方面,高阶突变体往往比低阶突变体展现出更高的活性。
PROTLGN 模型能够成功地识别高增益功能突变体,并且在组合单突变位点时能够识别出正向的上位效应。这证实了 PROTLGN 在指导 Ago 蛋白突变设计中的有效性,尤其是在提高抗体的性能方面。
将 PROTLGN 与其他自监督模型比较:更高效、更准确
在最新的研究中,科学家们利用 PROTLGN 模型对深度突变扫描 (DMS) 数据集中的蛋白质适应度进行了预测,并与其他自监督学习模型进行了比较。
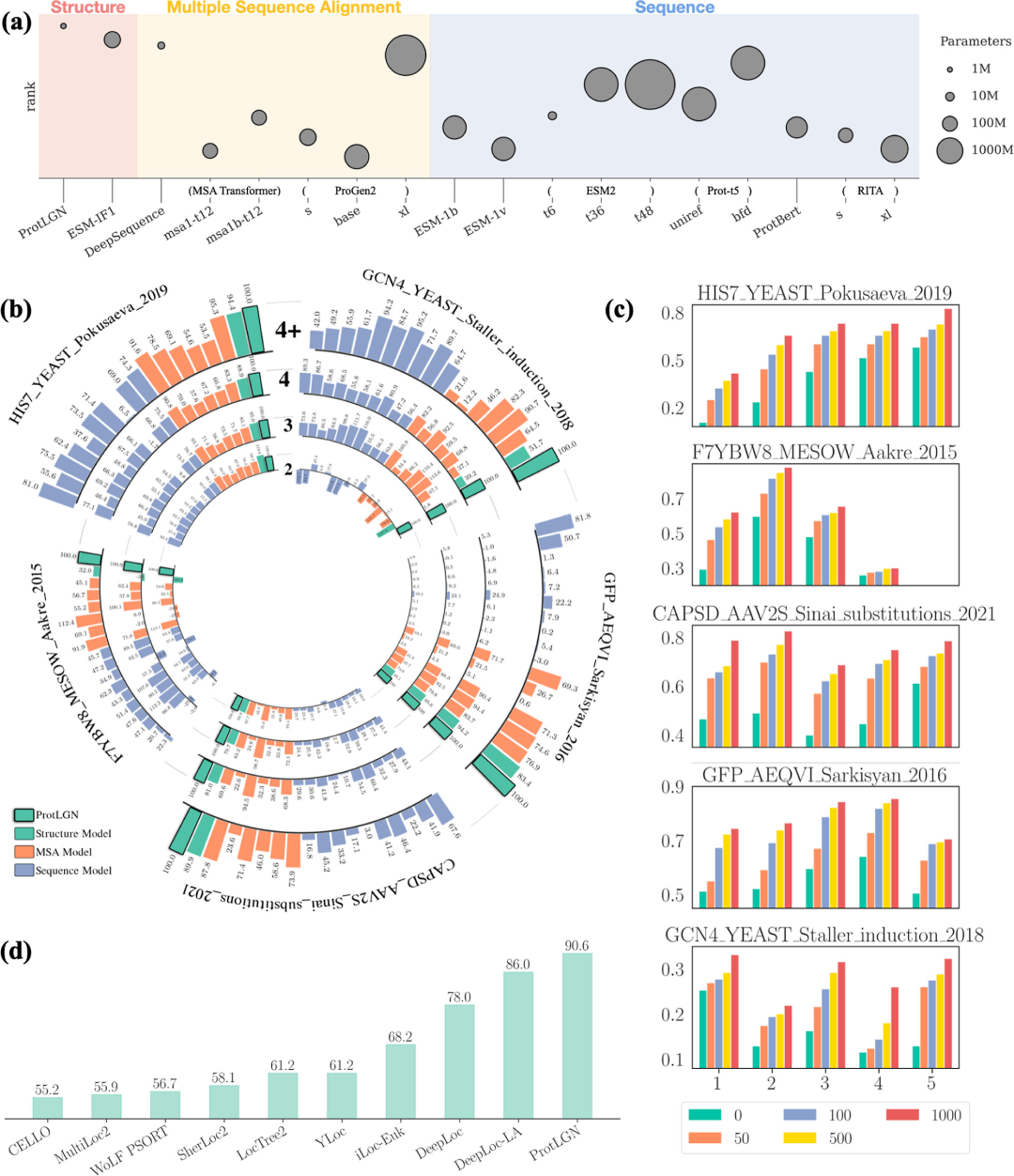
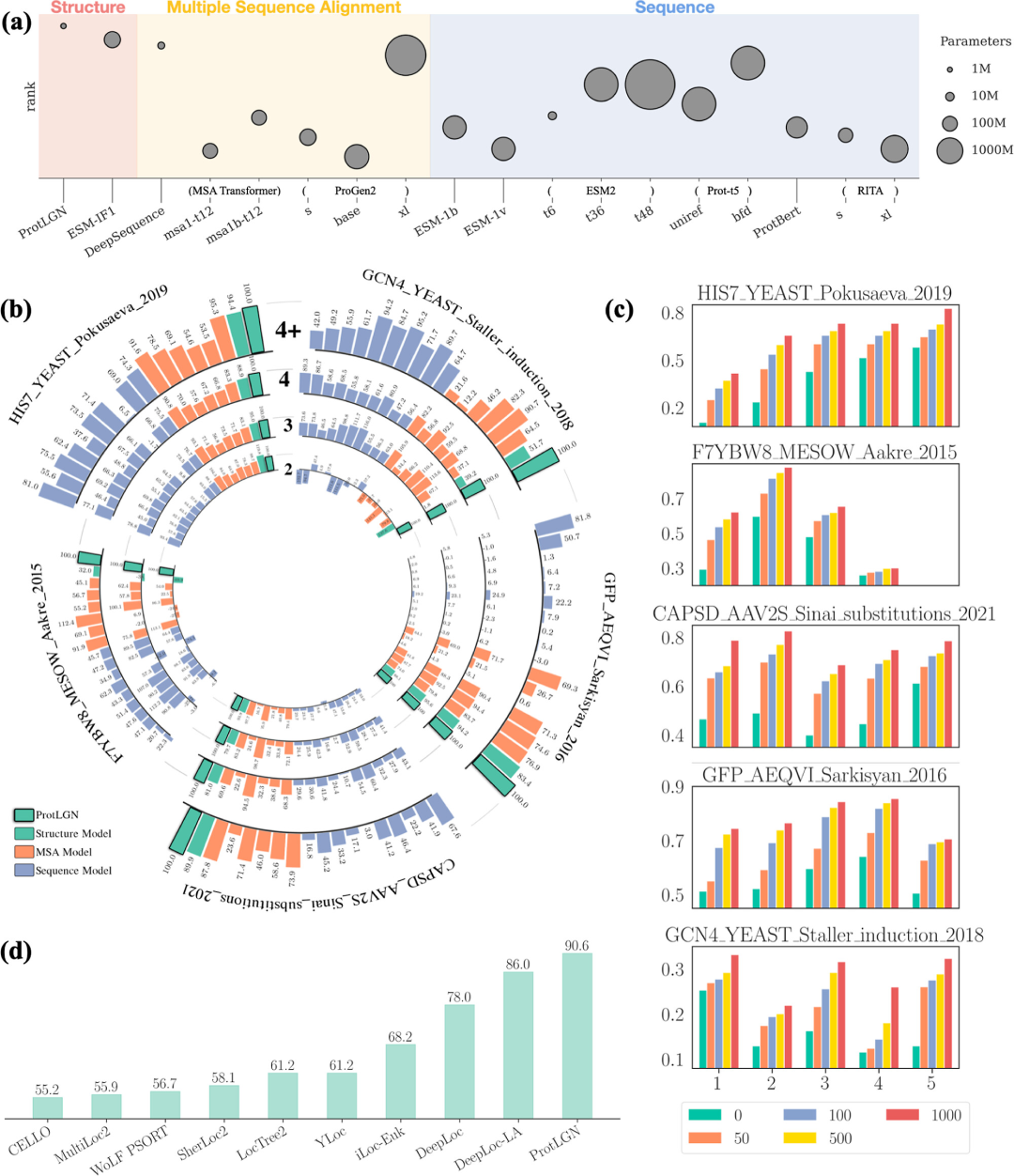
不同模型的蛋白质预测效果
a:零样本深度学习模型的推断效率和效果
b:多突变位点效应预测性能
c:高阶突变预测性能提升
实验结果显示,PROTLGN 在所有比较的模型中表现最佳,它不仅准确预测了蛋白质的适应度,而且使用的可训练参数数量最少。这一点非常重要,因为较少的参数意味着模型训练和微调的成本低,同时也意味着模型可以在较少的标记数据上进行有效学习。
在实验的最后阶段,研究人员使用了部分可用的实验标签来增强模型的微调,进一步提高了预测的准确性,结果显示 PROTLGN 在性能上明显优于其他方法,尤其是在处理高阶突变体时。
PROTLGN 对蛋白质亚细胞定位预测:综合分析蛋白质的三维结构
在一项突破性的研究中,科学家们采用了 PROTLGN 模型来预测蛋白质的亚细胞定位 (protein subcellular localization, PSL),即蛋白质在细胞内的具体位置,它与蛋白质的功能密切相关。
模型对蛋白质亚细胞定位预测
研究团队首先利用 PROTLGN 模型分析了 9,366 个标记的蛋白质,每个蛋白质由其氨基酸级别的表示组成。随后,在 2,738 个测试蛋白质上进行了评估,以预测这些蛋白质在细胞内的 10 个可能位置。实验结果显示,PROTLGN 在预测准确性上显著超越了现有的基于氨基酸序列或同源信息的基线方法。
结语:生物医学的「AI 革命」没有边界
从 AlphaFold 开始,人工智能不断刷新着生物医学工程的认知界限,但深度学习仍受限于高质量的数据,对于这一局限,PROTLGN 的零样本学习训练或许给出了回答。零数据跨入 AGI 时代后,下一代结构生物学家很可能不再主要是实验方法的专家,更多地是负责解释、设计和执行基于结构的实验,证明或否定生物学中的机制,或设计新的蛋白质功能与临床治疗方法。
来源: HyperAI超神经


 科普中国公众号
科普中国公众号
 科普中国微博
科普中国微博

 帮助
帮助
 HyperAI超神经
HyperAI超神经 
