出品:科普中国
作者:冉浩(中国科学院昆明动物研究所生物多样性基因组学课题组) 周龙(浙江大学生命演化研究中心)
监制:中国科普博览
在生物学领域,人类属于一个被称为灵长类的哺乳动物类群。类人猿、猴类……在灵长类动物中,人类又拥有众多“亲戚”,我们和它们拥有共同的祖先,那么这个共同的祖先究竟是谁?我们是怎样一步步变成人类的?这些灵长类动物的起源问题一直为人们所关注。近日,随着灵长类基因组计划阶段性成果的公布,我们离回答这个问题又进了一大步。
从祖先到枝繁叶茂
要回答“我们是怎样一步步变成人类的”这个问题,就要弄明白我们人类和灵长类“亲戚”之间错综复杂的演化关系,灵长类基因组计划就为我们打开了灵长类演化的“黑匣子”。
浙江大学的张国捷课题组和中国科学院昆明动物研究所的吴东东课题组,联合国内外学者,利用50种灵长类动物的全基因组数据进行了溯祖分析(也就是反推灵长类各个类群的起源)。
这项工作需要样本对生物类群有较高的覆盖度,才能获得比较准确的结果。此次研究的样本涵盖了14个科共38个属的样本,其中加入了27个新的高质量基因组数据,还包括了之前研究中较少涉及的种群。
因此,这项研究在结果的可靠度和物种的覆盖度上比之前的研究有了较大的提升。
灵长类样本选择完毕后,研究团队又以鼯猴和树鼩这两种与灵长类关系比较近但并非灵长动物的基因组作为外群,重构了灵长类的演化关系,并得到了各主要类群之间在历史上彼此朝不同方向演化的发生时间。
结果如何?这要从灵长类这个庞大的“家族”说起。
灵长类动物有超过500个现生物种,生物分类学家将他们归入到了灵长目共计16个科的79个属中。
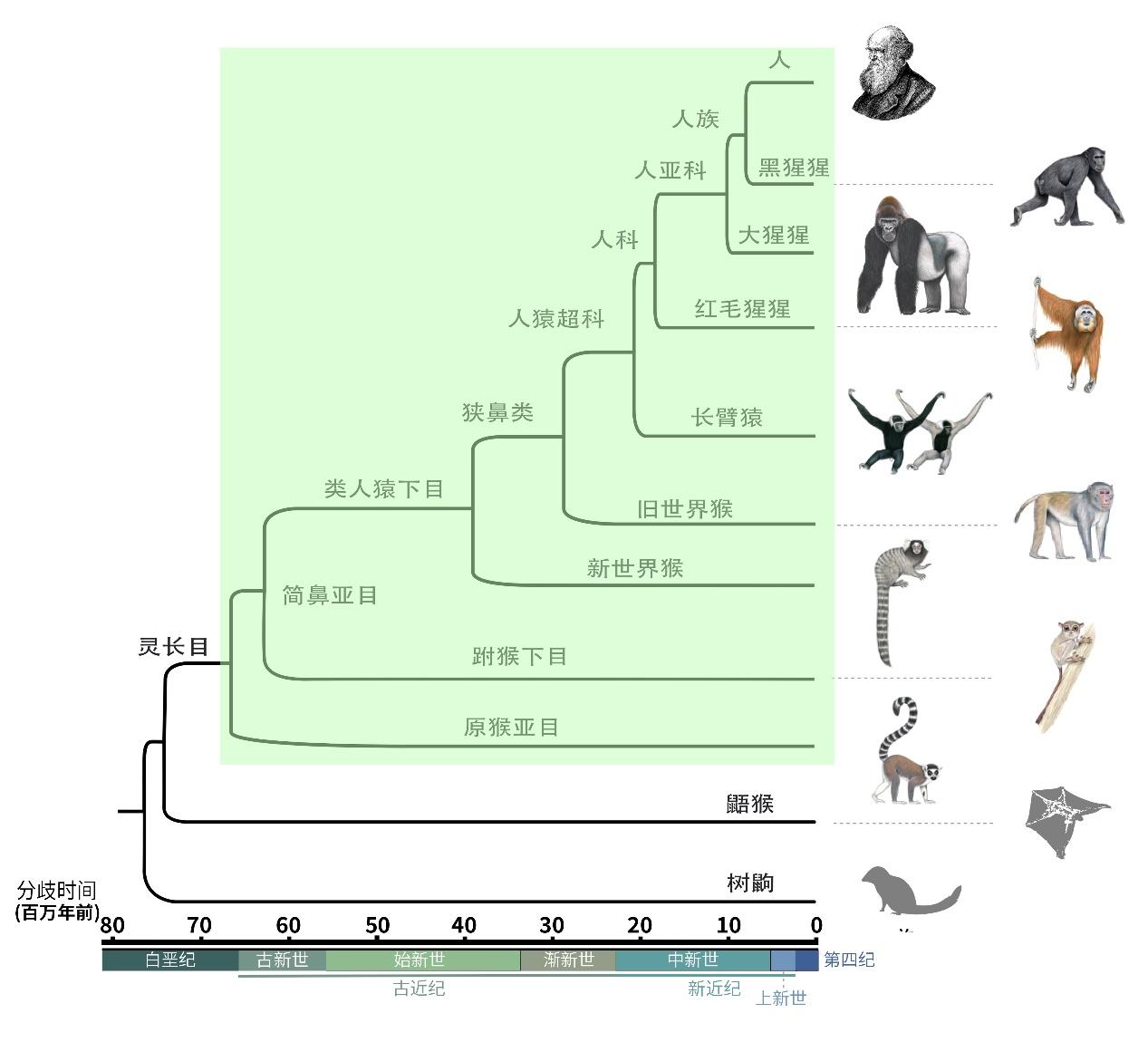
灵长目演化树
(图片来源:灵长类图片由Stephen D. Nash绘制,图片由张国捷课题组和吴东东课题组合作提供)
其中,原猴类(原猴亚目,Strepsirrhini)是比较原始的类群,它们分布在非洲、南亚和东亚,狐猴、懒猴和丛猴等就属于这个类群。而现代灵长类的主体简鼻类(简鼻亚目,Haplorrhini)则又包括了狭鼻类(Catarrhini)和阔鼻类(Platyrrhini)两个大类,后者也是两个不同的演化分支。
狭鼻类主要分布在欧亚大陆和非洲大陆,比较直观的形态特征之一是两个鼻孔的间距很短,我们人类就属于狭鼻类(你可以摸摸自己的鼻子感受一下这个特征)。狭鼻类中的猴类被习惯性地称为旧世界猴(按照传统说法,欧亚非大陆属于“旧大陆”,美洲大陆则是“新大陆”),包括了我们常说的狒狒、猕猴、叶猴等等。
狭鼻类的另一支——猿类,是旧世界猴的姐妹群,也分布在“旧大陆”。诗词“两岸猿声啼不住”中的长臂猿属于小猿,而黑猩猩、红毛猩猩、大猩猩和人类等则属于大猿,在一些刻意强调我们出身的场合,有时我们也会自称“裸猿”。人类在从大猿中演化出来的过程中,失去了浓密的体毛,获得了更加彻底的直立行走姿态、更加发达的大脑结构以及更加复杂的社会结构。
美洲大陆没有猿类,那里的灵长类动物是阔鼻类,也被称为新世界猴,新世界猴包括狨类、卷尾猴类、夜猴类、僧面猴类和蜘蛛猴类等。顾名思义,它们的鼻孔间距很大。此外,新世界猴的尾巴非常灵活,可以辅助攀爬。
这项研究分析得出了所有灵长类的最近共同祖先可能出现在大约6829万—6495万年前。这个时间段,距离6550万年前那次造成非鸟恐龙灭绝的白垩纪末期大灭绝事件非常近,大致处于白垩纪的时间边界位置。这意味着灵长类动物的演化可能受到了物种大灭绝事件的影响,至于如何受到影响的,那就是未来的研究需要回答的问题了。
快速演化的大脑
从猿到人,大脑的变化非常关键。
人类可能拥有动物界最聪明的大脑,不仅脑容量较大,大脑皮层的结构也更为复杂。和身体的其他部分一样,人类的大脑也是从灵长类的祖先那里演化而来的。
在漫长的演化过程中,灵长类的脑部容量逐渐增加,占身体的比例也逐步增加,且皮层折叠程度不断复杂化。在灵长类的演化过程中,相对脑容量的显著增大有四个关键节点,分别发生在类人猿下目的祖先、狭鼻类祖先、大猿祖先和人类身上。
在猩猩等大猿物种出现之后,这种趋势变得尤其突出,并在人类中达到了顶峰。人类不仅拥有了灵长类动物中最大的脑容量,还拥有了折叠程度最为复杂的大脑皮层。

灵长类物种脑容量演化历程以及此过程中基因组上的变化
(图片来源:大脑图像来自密歇根州立大学,图片由张国捷课题组和吴东东课题组合作提供)
研究发现,在灵长类演化的过程中,很多与大脑发育相关的基因在演化的关键节点受到了正向选择,也就是说,这些基因被自然选择强化了。因此,研究人员推测,这些基因对灵长类脑容量的增大发挥了重要作用。
这些基因中还包括一些前人的实验研究已经发现的、与大脑发育相关的关键基因,这些基因的突变会导致小鼠的大脑功能受损。例如,小头畸形是人类严重的神经系统缺陷,病人脑容量由于神经细胞增殖的能力受到阻断而变小。与小头畸形相关的基因在多个分支中都受到了强烈的正向选择,很可能在灵长类的脑容量增大过程中发挥了作用。
此外,研究人员还发现,一些非编码区域在灵长类演化过程的关键节点发生了加速演化。非编码区域是一些不会表达出蛋白质的DNA区域,但是这一区域会影响基因表达,如最终增加或减少相应蛋白质的合成量。这些区域很多落在大脑发育相关基因的调控区域,这些结果表明了灵长类动物在漫长的演化过程中,通过调节大脑相关基因的表达不断地优化大脑的构造。
以上这些发现表明,**灵长类动物在演化历程中,最终演化成大脑更为发达的形态,有很多基因及其调控区域参与其中,**这丰富了我们对灵长类大脑演化分子机制的认识。
寻找人类失去的尾巴
在演化过程中,人类丢掉了身后的尾巴。
不论长短,几乎所有的脊椎动物都有尾巴,在屁股上长的尾巴也叫肛后尾。对于一些动作灵敏的灵长类物种,长短不一的尾巴能够帮助它们稳定身体,转向和控制速度,阔鼻猴的尾巴还能起到辅助攀爬的抓握作用。然而值得注意的是,猿类的祖先却失去了尾巴,这也成为了猿类的重要特征。猿类是如何失去尾巴的呢?
有意思的是,猿类的尾巴消失得非常彻底,直到目前也没有猿类祖先逐渐失去尾巴的化石记录。早期猿类化石——距今约2000万年前的原康修尔猿(Proconsul),就已经没有了尾巴。猿类失去尾巴获得了直立行走的优势,同时也丧失了攀爬时用尾巴保持平衡的功能,这可能促进了部分猿类从树冠走向地面。
此前,已有生物学家对此做出了一些探讨,从适应性方面推测了猿类失去尾巴的原因。在分子生物学层面上,相关研究还非常有限,推测这一现象可能与一些特定的基因调控序列的突变有关。
在此次研究中,通过检测人猿超科物种的基因组相对于其他灵长类发生的变化,研究人员发现,多个基因的非编码调控区域积累了大量变异,比如基因KIAA1217的调控区域。
KIAA1217基因在人类身上发生突变可能会导致脊柱和尾椎畸形,影响脊柱的正常发育;而在小鼠中,这个基因的突变则会导致尾椎数量的减少。基因调控区域是DNA上能够调控基因发挥作用的特殊区域,或者说,是一些特殊的DNA序列。这个基因调控区域在基因KIAA1217的增强子区域,并且与基因在同一个拓扑结构关联域(TAD)中。这提示着,这个基因调控区域和基因有很强的交互作用,可能调控了KIAA1217基因的表达。
猿类的这个基因调控区域的DNA序列和其他灵长类有很大不同。研究人员推测,很可能是这些区域的突变导致了KIAA1217基因表达失衡,导致猿类失去尾巴。尽管这一假说仍需进一步的研究和验证,但是这一发现已经为我们更好地理解猿类演化史提供了新的线索。
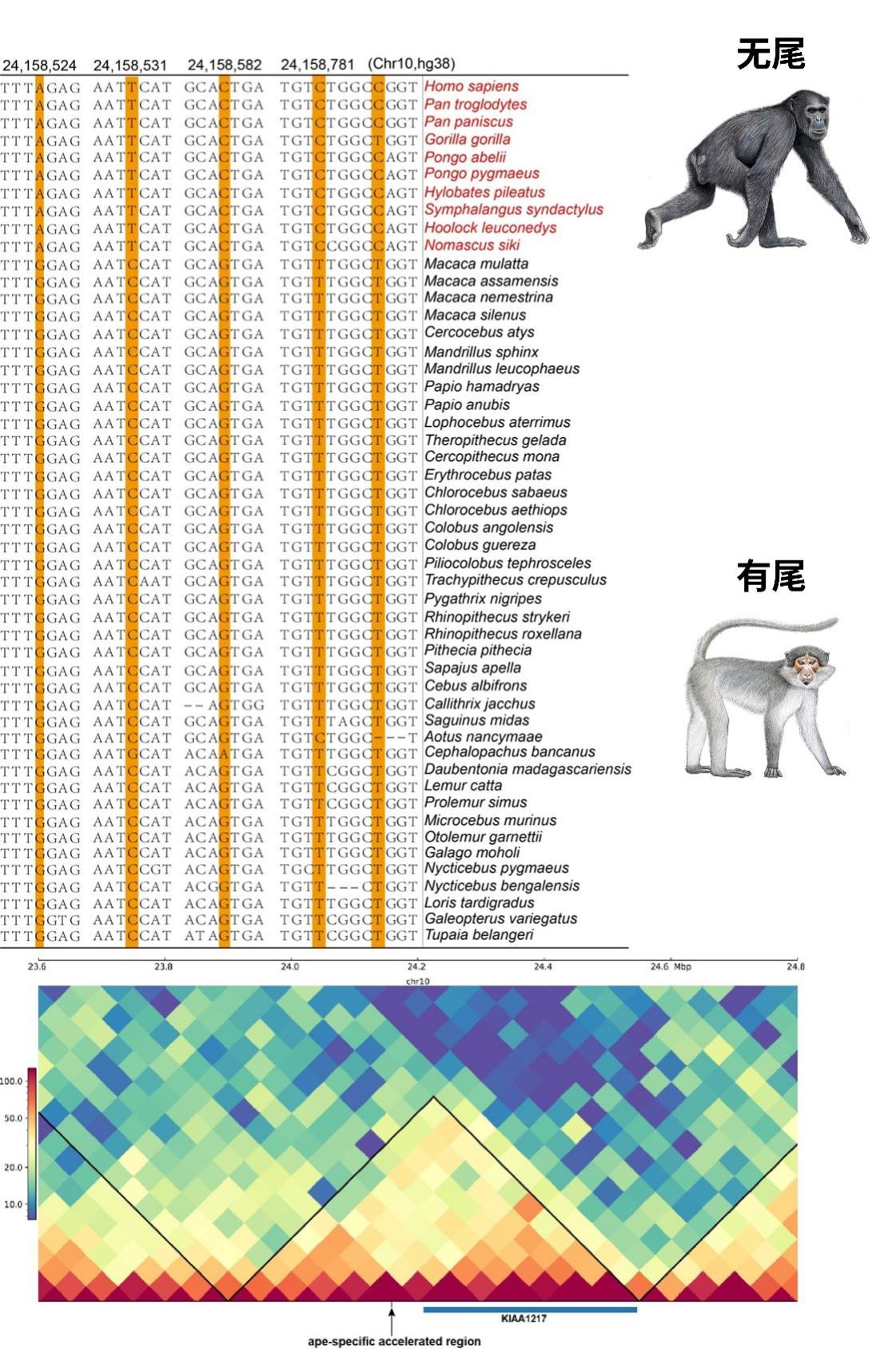
KIAA1217基因的调控区域在猿类中的快速演化可能导致其丢失尾巴
(图片来源:张国捷课题组和吴东东课题组合作供图)
骨骼、体型、消化系统……
在演化过程中,灵长类动物的共同祖先在适应着各种环境和食物的同时,也不断地经历着骨骼、体型和消化系统的演变。除了大脑的快速演化,这些方面的演化也对灵长类的适应性和生存能力有着重要的影响。本次研究还发现了许多重要的基因与这些变化相关。
骨骼系统在灵长类动物演化过程中的作用异常突出,骨发育相关的基因在树栖生活方式的适应性演化中扮演着特别重要的角色。在灵长类祖先中,有四个与骨发育相关的基因(PIEZ01, EGFR, BMPER and NOTCH2)受到了强烈的正选择,具体功能有待进一步明确。研究人员在长臂猿中也发现了四个正选择基因(LONP1, BRCA2, NEK1 and SLC25A24),这些基因的变异会影响骨骼长度,从而延长前臂,并且对长臂猿在树上活动和觅食等方面产生重要作用。
灵长类动物的体型变化很大,小如只有几十克的鼠狐猴,大有超过200千克的大猩猩。研究者在大猿祖先的基因中发现了几个重要基因,可能对大猩猩体型的演化产生了影响。其中一个是DUOX2基因,它参与对身体发育有重要作用的甲状腺激素的合成。DUOX2基因变异能导致小鼠体型变小。此外,还有一些基因参与塑造了骨骼发育和体型大小的调节通路。
不同的灵长类有不同的饮食习惯和对应的消化系统,有些灵长类(如叶食性的疣猴类)很喜欢吃树叶,并且已经演化出了独特的前肠系统以适应这类饮食。研究发现了一些关键的消化基因,这些基因在疣猴类的祖先身上受到了正选择,从而积累了特殊的氨基酸变异,来适应这种特殊饮食的状态。
例如,ACADM基因编码一种在代谢摄入的脂肪酸方面起着关键作用的Acyl-CoA脱氢酶,疣猴在这个基因上发生的变化提高了它们消化脂肪酸的能力。再例如,累积的NOX1基因变异可以进一步帮助疣猴调节体内的微生物,让它们更好地消化叶子。它们的肠道还可以通过微生物发酵,产生短链挥发性脂肪酸,从而提供更多的能量。
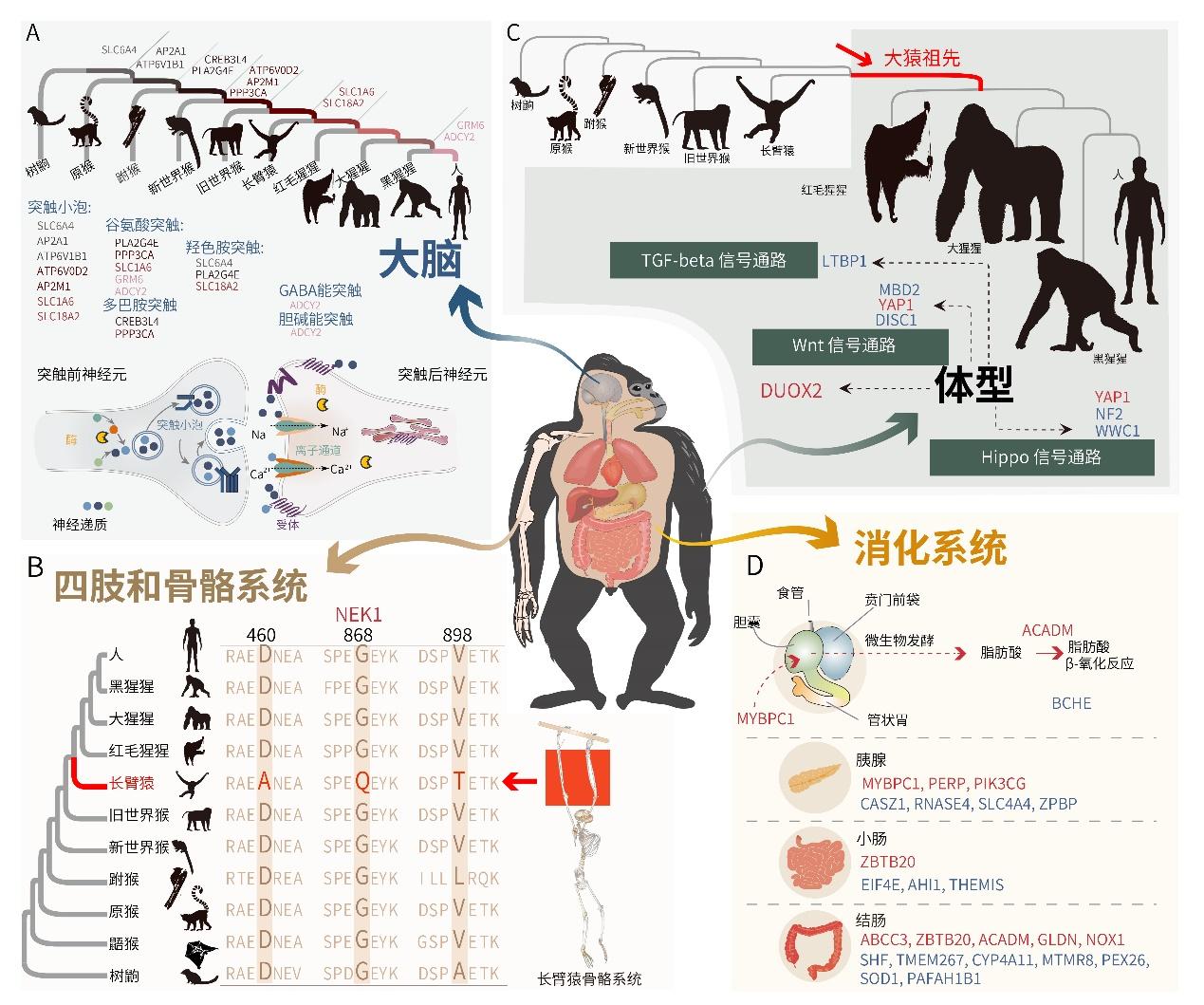
灵长目基因组演化特征与表型特征之间的关联
(图片来源:张国捷课题组和吴东东课题组合作供图)
结语
灵长类基因组计划不仅探讨了包括猿类在内的灵长类的演化历程和物种形成(如首次报道了灵长类的杂交成种现象),还对染色体演化、快速演化DNA序列、基因的不完全谱系分流等等进行了探讨,它的成果远远不止这些,要知道,这是一项庞大又深入的研究。
在寻找生命起源的旅途中,生物学家们从不会因为畏难而止步不前。我们究竟是谁?为了回答这个问题,人类在一组组字母中“解谜”,探求演化的奥秘。我们坚信,终有一天,人类会书写出完整的生命故事。
注:
灵长类基因组计划介绍
全球现存有超过500个灵长类物种,分属于16科9属。非人灵长类由于其与人类的近缘关系,对于理解人类的起源、演化、生理性状、疾病等方面的研究具有重要意义。我国科学家联合国内外多个研究中心,发起了灵长类基因组计划,旨在通过多学科交叉技术手段和团队合作,研究人类在内的灵长类物种的起源和分化过程,以及灵长类社会组织和各种生理特征的演化和遗传基础。此外,该计划还将研究灵长类遗传变异图谱及其对人类致病基因变异模式的影响。
灵长类基因组计划的研究联盟
浙江大学生命演化研究中心张国捷教授团队
中国科学院昆明动物所吴东东研究员团队
西北大学生命科学学院齐晓光教授团队
云南大学生命科学学院于黎研究员团队
西班牙庞培法布拉大学联合演化生物学研究所Tomàs Marquès-Bonet教授团队
Illumina人工智能实验室
美国贝勒医学院人类基因组测序中心Jeffrey Rogers教授团队
丹麦奥胡斯大学Mikkel H. Schierup团队
德国莱布尼茨灵长类研究所Christian Roos教授团队
阶段性研究成果(11篇研究论文)
Science研究专刊(8篇论文)
Fiziev PP, Mcrae J, Ulirsch JC, ..., Farh K K-H. 2023. Rare penetrant mutations confer severe risk of common diseases. Science 380. Doi: 10.1126/science.abo1131.
Gao H, Hamp T, Ede J, ..., Farh K K-H. 2023. The landscape of tolerated genetic variation in humans and primates. Science 380. Doi: 10.1126/science.abn8197.
Kuderna LFK, Gao H, Janiak MC, ..., Bonet TM. 2023. A global catalog of whole-genome diversity from 233 primate species. Science 380, 906-913. Doi: 10.1126/science.abn7829.
Qi XG, Wu JW, Zhao L, ..., Li BG. 2003. Adaptations to a cold climate promoted social evolution in Asian colobine primates. Science 380.Doi: 10.1126/science.abl8621.
Rivas-gonzález I, Rousselle M, Li F,..., Zhang GJ. Pervasive incomplete lineage sorting illuminates speciation and selection in primates. Science 380.DOI: 10.1126/science.abn4409.
Shao Y, Zhou L, Li F,..., Wu DD. 2023. Phylogenomic analyses provide insights into primate evolution, Science 380, 913-924.Doi: 10.1126/science.abn6919.
Sørensen EF, Harris RA, Zhang LY, ..., Rogers J. 2023. Genome-wide coancestry reveals details of ancient and recent male-driven reticulation in baboons. Science 380. Doi: 10.1126/science.abn8153.
Wu H, Wang Z, Zhang Y, ..., Yu L. 2023. Hybrid origin of a primate, the gray snub-nosed monkey. Science 380. Doi: 10.1126/science.abl4997.
Science Advances(2篇)
Bi XP, Zhou L, Zhang JJ, ..., Zhang GJ. 2023. Lineage-specific accelerated sequences underlying primate evolution. Science Advances 9. Doi: 10.1126/sciadv.adc9507.
Zhang BL, Chen W, Wang ZF, ..., Wu DD. 2023. Comparative genomics reveals the hybrid origin of a macaque group. Science Advances 9. Doi: 10.1126/sciadv.add3580.
Nature Ecology & Evolution(1篇)
Zhou Y, Zhan XY, Jin JZ, ..., Zhang GJ. 2023. Eighty million years of rapid evolution of the primate Y chromosome. Nature Ecology & Evolution. Doi: 10.1038/s41559-022-01974-x.
(注:文中拉丁文部分应为斜体。)

来源: 中国科普博览
内容资源由项目单位提供


 科普中国公众号
科普中国公众号
 科普中国微博
科普中国微博

 帮助
帮助
 中国科普博览
中国科普博览 
