精准调控特定细胞的基因表达对于基因治疗、合成生物学等领域的进展至关重要,此过程依赖于被称为「顺式调控元件(Cis-Regulatory elements, CRE)」的一类 DNA 序列,如启动子(Promoter)、增强子(Enhancer),它们就像基因的「开关」一样,决定基因在目标细胞中「启动」或「关闭」,同时避免在其他正常细胞中异常激活。然而,自然存在的有效 CRE 数量有限且难以精准匹配多样化的生物医学应用场景。更关键的是,DNA 序列的可能性呈指数级增长,如 100 个碱基的序列就有 4¹⁰⁰ 种组合,想要通过实验逐一验证可谓难如登天,不仅费时费力还无法满足实际需求。
当前基于深度学习的方法已经大幅提升了实验效率,但现有方法仍面临多重挑战。例如,部分方法通过突变现有 DNA 或随机序列优化,容易陷入「局部最优」的陷阱,导致生成的有效序列多样性不足;基于自回归语言模型(Autoregressive Language Models)的方案虽能捕捉 DNA 序列规律,但仅能「模仿已知序列」,难以探索全新的细胞特异性 CRE;基于强化学习(Reinforcement Learning, RL)的方法虽然提升了目标细胞的调控效果,却忽略了对其他细胞的「副作用」控制。此外,这些标准设计框架还往往忽视了对生物学合理性的考量,生成的序列可能因未匹配关键转录因子结合位点(Transcription Factor Binding Sites, TFBSs),进而导致实际调控功能失效。
为了填补细胞特异性 CRE 精准设计的空白,多伦多大学团队联合昌平实验室等机构开发了一种名为 Ctrl-DNA 的约束强化学习框架。该框架以预训练的 DNA 语言模型为基础,通过强化学习算法在优化过程中同步实现「双目标」:一方面最大化 CRE 在目标细胞中的调控活性,另一方面又严格限制其在非目标细胞中的活性。同时,采用拉格朗日乘数数学工具平衡双重需求,并参考真实 DNA 中的 TFBS 分布,确保生成序列的生物学有效性。
研究结果表明,在 6 种人体细胞的设计任务中,Ctrl-DNA 生成的 CRE 在「目标细胞类型中的高活性」和「非目标细胞类型中的约束度」两大关键指标上显著优于现有方法,且保持了显著的多样性,为合成生物学「造可控系统」、基因治疗「避脱靶风险」、精准医学「做细胞级定制」提供了全新解决方案。
相关研究成果以「Ctrl-DNA: Constrained Reinforcement Learning for Cell-Specific Cis-Regulatory Element Design」为题,发表于 arXiv 预印本平台,并入选 NeurIPS 2025。
研究亮点:
* 提出全新约束感知强化学习框架,为设计 CRE 进行精确的细胞类型特异性基因表达提供了工具
* 简化优化流程并提升了实验效率、降低了计算成本
* 实验验证了 Ctrl-DNA 兼具功能有效性与生物学合理性

论文地址:
https://arxiv.org/abs/2505.20578
关注公众号,后台回复「 Ctrl-DNA」获取完整 PDF
更多 AI 前沿论文:
https://hyper.ai/papers
数据集:基于真实的人类启动子、增强子数据集
在本次研究中,研究人员采用了真实的人类启动子和增强子数据集对 Ctrl-DNA 进行评估验证。
其中,人类启动子数据集包含来自三种白血病衍生细胞系的启动子活性数据,这三种细胞系分别是:Jurkat 细胞系、K562 细胞系和 THP1 细胞系。这三种细胞系均为中胚层衍生的造血细胞系,具有高度的生物学相似性。该数据集的每条序列长度为 250 碱基对。如下表所示:
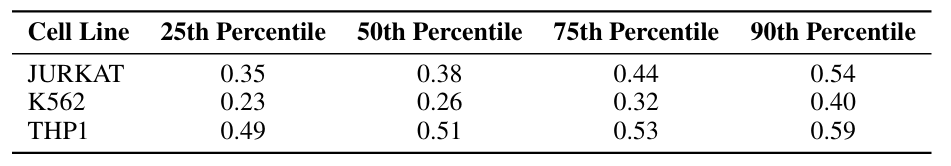
人类启动子数据集(标准化活性得分百分位统计)
人类增强子数据集包含通过大规模平行报告实验(MPRA)测得的、来自三种细胞系的 CRE 活性数据,这三种细胞系分别为:HepG2(肝细胞系)、K562(红细胞系) 和 SK-N-SH (神经母细胞瘤细胞系)。该数据集的每条序列长度为 200 碱基对。如下表所示: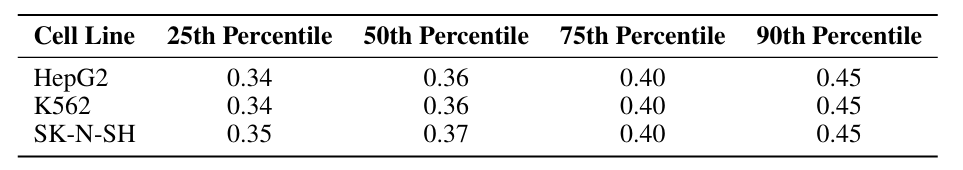
人类增强子数据集(标准化活性得分百分位统计)
值得注意的是,在 THP1 细胞系中,第 25 百分位活动达到了 0.49,呈现右偏分布。这种分布偏差可能是导致 THP1 细胞系活性约束难度增加的部分原因。
模型架构:基于预训练 DNA 语言模型,合并拉格朗日松弛
Ctrl-DNA 作为一种基于约束强化学习的调控 DNA 序列设计框架,其核心目标是生成具有可控细胞类型特异性的 CRE。
在功能实现上,它既要最大化目标细胞中 CRE 的适配度,即提升基因表达增强能力,又需要将脱靶细胞中的适配度严格控制在预设阈值内。与此同时,它还要确保生成序列符合真实生物学规律,避免出现「实验达标应用无效」的情况。
为此,研究人员从框架易用性、合理性等多方面进行了考量,对框架构建进行了详细的设计。如下图所示: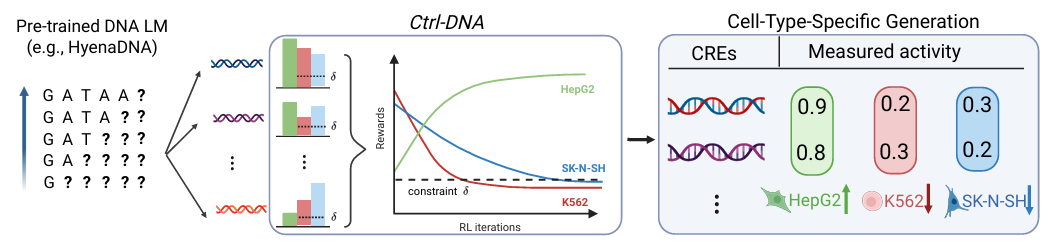
Ctrl-DNA 框架概述
在模型与输入方面,Ctrl-DNA 以在人类基因组上预训练 HyenaDNA 自回归基因组语言模型为初始策略模型进行微调,同时采用 Enformer 架构训练细胞类型特异性奖励模型,结合大规模平行报告实验测得的「序列-适配度」数据,分别计算目标细胞奖励与脱靶细胞奖励。
在问题建模层面,研究人员将 DNA 序列设计转化为了约束马尔可夫决策过程(constrained Markov decision process,CMDP)。Ctrl-DNA 核心的优化机制采用约束批处理相对策略优化(Constrained Batch-wise Relative Policy Optimization),该机制通过拉格朗日松弛(Lagrangian Relaxation)将约束优化问题转化为无约束的原始对偶优化问题。优化过程通过迭代实现,策略更新以学习率沿拉格朗日目标函数梯度上升,脱靶细胞奖励的约束则通过调整拉格朗日乘数实现——对超出阈值的脱靶细胞增加拉格朗日乘数以强化约束,对满足阈值的脱靶细胞降低拉格朗日乘数以弱化约束。
为了降低训练复杂度,Ctrl-DNA 摒弃了传统强化学习中对价值模型的依赖,直接基于批数据统计计算归一化优势,以此引导策略优化选择「高目标奖励+低脱靶奖励」的序列。
在策略更新目标函数设计上,研究人员采用了「裁剪替代目标」+「KL 正则化」的组合,通过裁剪限制策略突变,同时引入当前策略与初始参考策略的 KL 散度,确保生成序列与天然 DNA 模式的一致性,最终形成策略更新目标函数。
为进一步保障生物学合理性,Ctrl-DNA 引入 TFBS 频率相关性作为额外约束。首先从真实高特异性 CRE 序列中,通过 FIMO 工具扫描 TFBS 并构建真实 TFBS 频率向量,再对每个生成序列计算其相应的 TFBS 频率向量,然后以皮尔逊相关系数作为额外约束奖励,同时将其对应的拉格朗日乘数裁剪至 [0,λmax](λmax ≤ 1),以平衡生物学合理性与目标优化,避免过度约束导致模型探索能力下降。
为保障模型训练稳定性,研究人员展示了实验中使用的超参数设置,所有模型都是用 Adam 优化器进行训练,策略学习率为 1e-4,批量大小为 256,训练周期为 100。实验在单个具有 40GB 内存的 NVIDIA A100 GPU 上进行训练。如下图所示: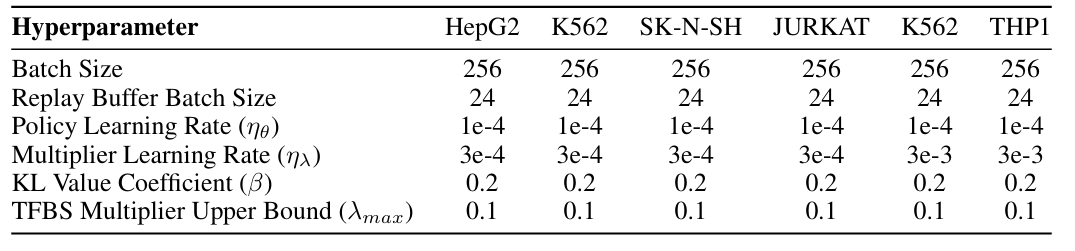
实验超参数
实验结果:与 8 类基线方法对比,Ctrl-DNA 优势凸显
Ctrl-DNA 的性能评估实验围绕人类增强子和启动子两大设计任务展开,覆盖上述提到的 6 种细胞系,并通过与进化算法(包括 AdaLead、Bayesian Optimization(BO)、CMA-ES、PEX)、生成模型(RegLM)以及强化学习方法(包括 TACO、PPO、PPO-Lagrangian)共 8 类基线方法对比,从细胞类型特异性、生物学合理性、序列多样性等多维度验证了其有效性和实用性。
在细胞类型特异性约束方面,Ctrl-DNA 展现出显著优势。如下图所示,横向坐标表示脱靶细胞类型的适应度;纵向坐标表示目标细胞类型的适应度。右上角所示方法表示实现了最大化目标细胞适应度和最小化脱靶表达之间的最佳平衡。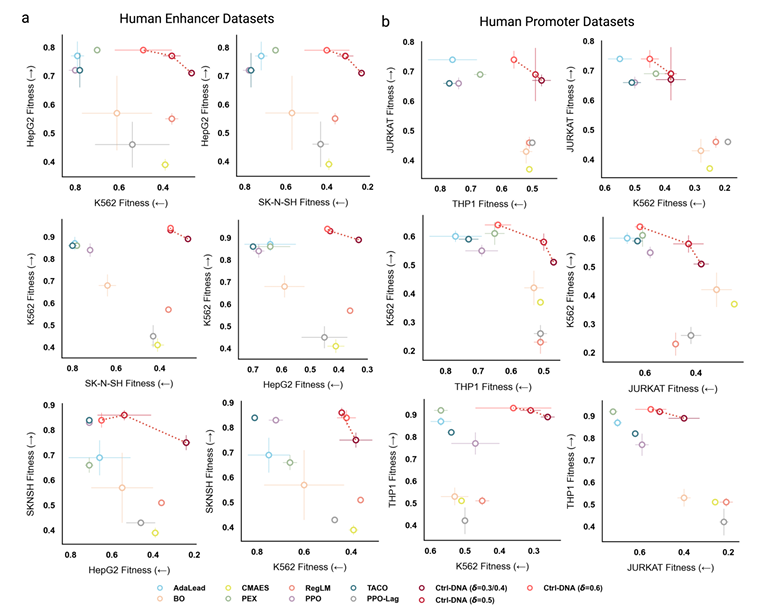
细胞类型特异性对比
对于增强子设计任务,在所有不同约束阈值(δ=0.3、0.5、0.6)下,Ctrl-DNA 在满足脱靶约束的同时始终达到最高的目标细胞适应度,即它均能在最大化目标细胞适配度的同时,严格满足脱靶细胞约束。另外 TACO 和 CMAES 等方法在目标细胞中虽获得了较高的表达,但它们无法抑制脱靶细胞的适应度,导致细胞类型特异性较差。
对于启动子设计任务,因为所有三种目标细胞类型都是中胚层来源的造血细胞,它们在转录方面有很大的相似性,为这项任务造成了较大挑战,但 Ctrl-DNA 依旧表现突出。实验设置了三个不同的约束阈值(δ=0.4、0.5、0.6)进行测试,Ctrl-DNA 在最大化目标细胞类型适应度和满足约束阈值 δ=0.5 和 0.6 时优于所有基线。另外值得注意的是,针对 THP1 细胞这类活性分布右偏的情况(上述数据集部分提到,25 百分位活性已达 0.49),没有任何一种方法能将其脱靶活性压至 δ=0.4 的严格阈值,但 Ctrl-DNA 已是所有方法中最接近约束要求的方法。
在生物学合理性验证中,如下图所示。Ctrl-DNA 在人类启动子和增强子的所有细胞类型中均实现了最高的奖励差异(△R),表明其更好地优化 DNA 序列的细胞特异性适应度。对于 motif 相关性,除 THP1 的启动子设计外,Ctrl-DNA 在大多数细胞类型中也取得了更强的性能。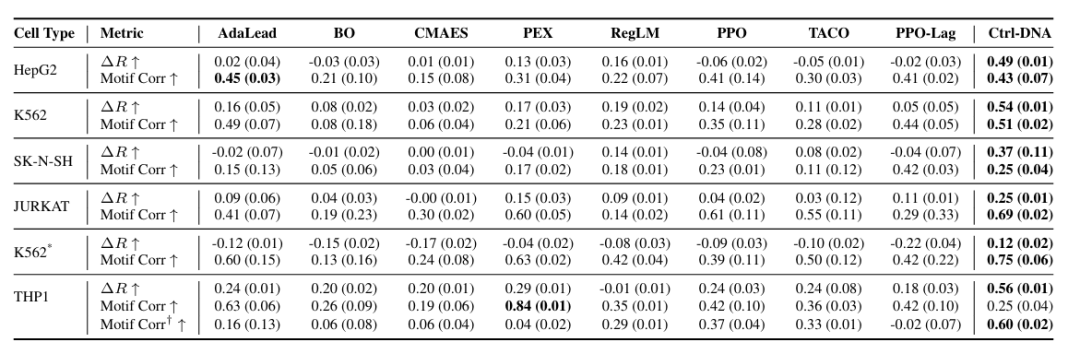
在人类增强子和人类启动子数据集上,针对每种目标细胞类型的不同方法的性能比较(阈值为 0.5)
为进一步探究这种差异,研究人员从 THP1 适应度的 90 百分位的启动子序列中提取 motif,并使用 q < 0.05 的阈值来避免假阳性,重新评估了生成的序列和参考集之间的 motif 相关性,如上图中表示为 motif Corr†。结果显示,Ctrl-DNA 在这种严格设置下依然优于所有基线,其相关系数提升至 0.60,而多数基线的相关性反而下降,说明其能优先捕捉具有功能意义的调控基序。
为进一步分析特定 TFBS 频率发现,研究人员特别地检查了 HepG2 肝细胞系和 K562 红细胞系特异性基序的生成序列。如下图所示,Ctrl-DNA 生成的 HepG2 序列显示了最高频率的肝脏特异性基序,如 HNF4A、HNF4G。同样的,为 K562 生成的序列包含最高频率的红细胞特异性基序,如 GATA1、GATA2。这表明,Ctrl-DNA 不仅优化了目标细胞的适应度,还学习了反映潜在细胞类型特异性的调控模式。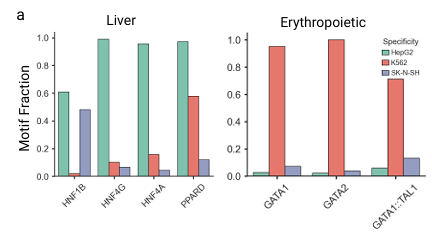
含有选定细胞类型特异性转录因子基序的 Ctrl-DNA 生成增强子的部分
在序列多样性方面,Ctrl-DNA 达到了与大多数基线相当或更高的多样性,证实了其在不牺牲调控控制的情况下生成多样化序列的能力。如下图所示: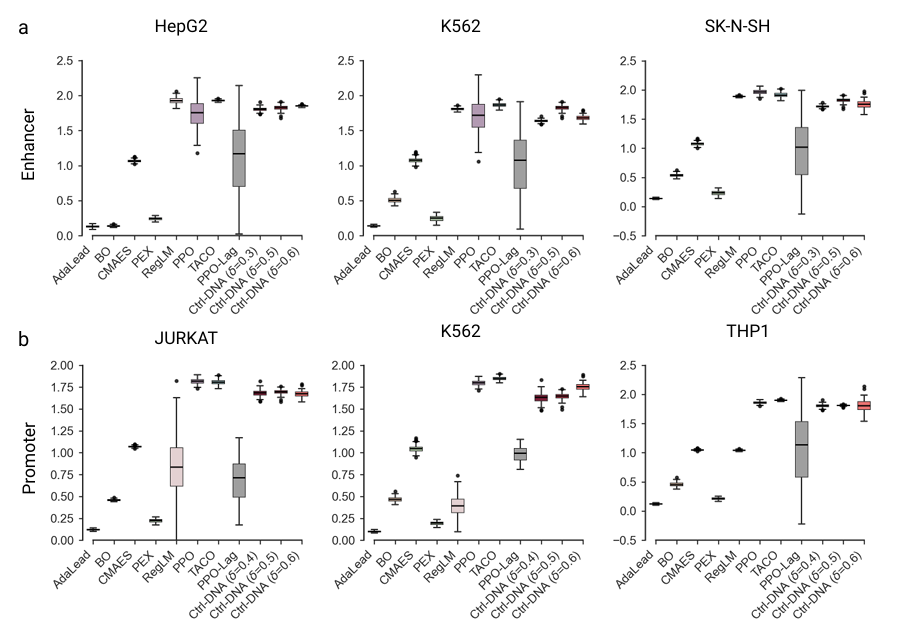
在人类增强子和启动子数据集上生成序列的多样性评分
最后,研究人员通过消融实验进一步验证了 Ctrl-DNA 核心模块的有效性。同时 TFBS 正则化模块的作用也得到证实,具有有效引导序列向生物学真实模式靠拢的作用。
AI 驱动 DNA 「开关」设计开启新篇章
过去,设计调控性 DNA 序列「开关」多靠「试错」,通过大量反复的手动筛选,而现在结合 AI 技术后,能通过算法预测「哪些 DNA 序列与目标调控蛋白匹配度最高」,大幅提升了设计效率和精准度,这也是 AI 驱动 DNA 开关设计成为新方向的核心原因,进而直接推动了基因治疗、合成生物学等领域从「粗放」走向「精准」。
而在这棵关于「AI 驱动 DNA 开关设计」的大树上, 本论文仅仅也只是其中一颗肥硕的果实,回归过往,不少实验室都已开展起相关研究。
例如,来自杰克逊实验室(The Jackson Laboratory)、布罗德研究所(Broad Institute)和耶鲁大学的团队曾在 nature 发布了题为「Machine-guided design of cell-type-targeting cis-regulatory elements」的研究。该研究利用人工智能设计出数千个新型 DNA 开关,这些开关可以精准控制基因在不同细胞类型中的表达。具体而言,研究人员构建了能准确预测 CRE 活性的深度卷积神经网络 Malinois,并开发了用于设计具有特定功能 CREs 的模块化平台 CODA,为开发报告基因、CRISPR 治疗、基因替代方法等提供了有力工具。
论文地址:
https://www.nature.com/articles/s41586-024-08070-z
除此外,还有上述文中提到的 RegLM,来自 Genentec 公司。在题为「Designing realistic regulatory DNA with autoregressive language models」的研究中,介绍了名为 RegLM 的框架,基于自回归语言模型结合监督序列-功能模型,设计具有特定属性的合成 CRE。同样的,RegLM 也是基于 HyenaDNA 框架,通过将功能标签编码为提示令牌并添加到 DNA 序列前缀,训练或微调模型以进行下一个令牌预测,从而生成具有所需功能的 DNA 序列,同时结合监督序列-活性回归模型筛选生成的序列。
论文地址:
https://genome.cshlp.org/content/34/9/1411.full#aff-1
总而言之,Ctrl-DNA 的开发无疑是 DNA 开关设计的又一步精进,虽然其仍旧存在一些问题或亟待改进的地方,诸如纳入额外的生物约束以进一步提高生成序列合理性和功能、调整拉格朗日乘数仍是经验之谈等,但这些工具的开发和精进无疑是为 DNA 开关设计开启了新的篇章,同时也在促进着人工智能与生物学交叉科学不断发展。
来源: HyperAI超神经


 科普中国公众号
科普中国公众号
 科普中国微博
科普中国微博

 帮助
帮助
 HyperAI超神经
HyperAI超神经 
